最近读了一篇发表在NeurIPS 2021上的文章,NeurIPS的全称是神经信息处理系统大会(Conference and Workshop on Neural Information Processing Systems),是全球最受瞩目的AI、机器学习顶级学术会议之一,位列CCF-A。模型是预测分子性质的,题目为Property-Aware Relation Networks for Few-Shot Molecular Property Prediction,基于属性感知的小分子属性预测关系网络,与之前的工作相比,提出了属性感知嵌入函数、自适应关系图学习模块,同时也使用了meta-learning, 比之前讲的Meta-MGNN效果要好。
原文传送门:https://papers.nips.cc/paper/2021/hash/91bc333f6967019ac47b49ca0f2fa757-Abstract.html
1. Introduction
1.1 背景
分子性质预测在药物发现中有重要作用,可以识别具有目标特性的候选分子。同时,分子性质预测也是一个小样本问题,因为标注数据少。目前有一些预测分子性质的方法,但是他们忽略了忽略了两个事实,一个是不同的分子性质归因于不同的亚结构,另一个是分子之间的关系因目标性质不同而不同。
通常,在分子性质预测领域使用机器学习方法 建立分子结构和特定特性之间的联系,它通常由两部分组成:一个分子编码器,将分子结构编码为固定长度的分子表示,一个预测器,它根据分子表示估计某个属性的活性。
为什么标记数据少:在药物发现的先导优化阶段,只有少量候选分子可以通过虚拟筛选进行评估;经过一系列实验室实验后,由于缺乏所需的特性,大多数候选药物最终无法成为潜在的药物,这些导致了标记数据较少。
2.2 本文工作
本文提出了一个属性感知关系网络(PAR)来处理上述问题,利用了 相关的子结构和分子之间的关系会随着不同的分子性质而变化 这个事实。
本文首先使用 属性感知嵌入函数,将通用分子嵌入 转换为 与目标属性相关的子结构感知空间;然后使用 自适应关系图学习模块 来联合估计分子关系图,并针对目标属性细化分子嵌入,从而可以在相似分子之间有效地传播有限的标签。
此外,本文还使用用元学习策略,在任务中选择性地更新参数,以便分别对 通用知识 和 属性感知 知识 进行建模。
2.模型介绍
2.1overview
① use a specially designed embedding function to obtain property-aware molecular embedding for each molecule
② adaptively learn relation graph among molecules which allows effective propagation of the limited labels
①用专门设计的embedding function获取每个分子的 属性感知 分子嵌入;
②自适应学习分子间的关系图,允许有限标签的有效传播
PAR的架构图如下:
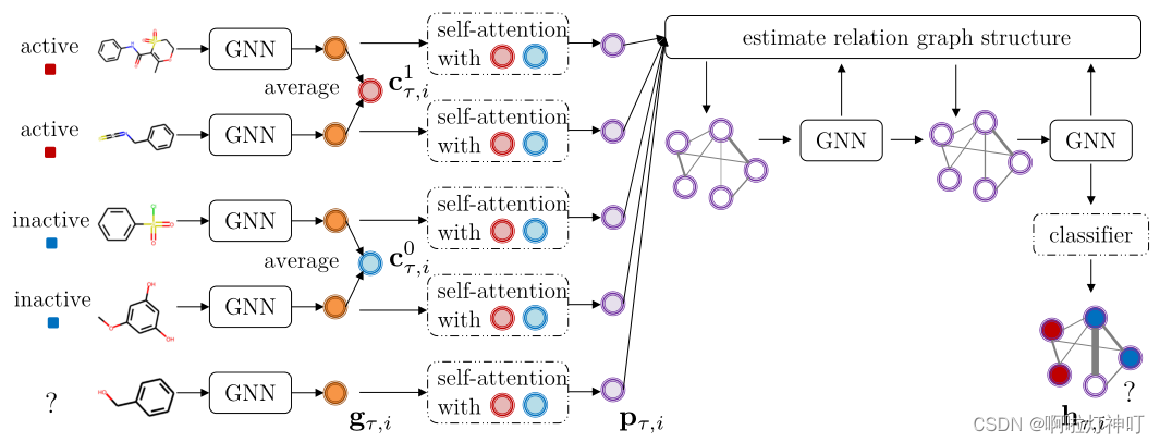
以 Tox21的2-way 2-shot 任务为例(其中一个task),2way是指这个性质是active(标签y=1)还是inactive(标签y=0),也就是有没有这个性质,2shot是每个类别有两个例子。
1.前部分是性质感知分子嵌入部分:
①首先使用基于图形的分子编码器(比如GNN)获 取分子嵌入g;
②然后使用 注意力机制 获取 性质感知分子嵌入p。
2.后部分是分子间自适应关系图:
①计算分子之间的相似性,得到相似性矩阵A;
②计算所有节点(分子)的嵌入h;
③预测分子性质。
目标是从一组小样本的分子性质预测任务中学习一个预测因子,并推广到预测 给定少量标记分子的 新性质。
训练时:实线的模块是固定的,虚线的模块在support set上进行微调。query中的分子XT,i 首先 使用基于图的分子编码器
表示为gT,i,然后使用 属性感知嵌入函数 表示为pT,i,进一步与关系图中分子在support
set中的嵌入共同适应为hT,i,作为最终的分子嵌入,用于类别预测。
2.2. Property-aware Molecular Embedding
由于不同的分子子结构具有不同的分子性质,本文设计了一个性质感知嵌入函数,将一般的分子嵌入 转化为 与 目标性质 相关的 子结构 感知空间。步骤如下:
2.2.1 step1:用基于图形的分子编码器获取分子嵌入gT,i
通过从大规模任务中学习,基于图形的分子编码器可以获得良好的分子嵌入,捕获分子共享的通用信息。因此,本文首先使用基于图形的分子编码器(如GNN和GCN等)来提取分子xT,i的嵌入gT,i,此时编码器的参数表示为Wg。
2.2.2 step2:获取性质感知分子嵌入pT,i
然而,现有的基于图的分子编码器无法捕获 对应属性的子结构(属性感知的子结构),尤其是在跨任务学习中 评估一个分子的多种属性时,分子和性质之间存在一对多的关系,这使小样本的分子性质预测特别困难。
因此,我们在嵌入空间中隐式捕获(关于属性预测任务TT的)目标属性的子结构。 cTc 表示类c∈{0,1} 的类原型,计算为:
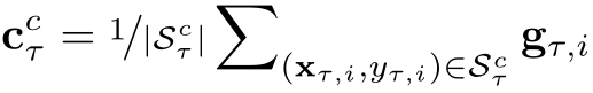
其中,
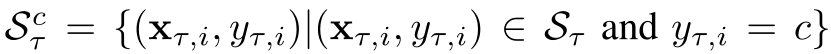
关于公式:比如说属于active的,也就是1的,让support中所有类为1的分子嵌入g求和,再除以总数,也就是取平均。Sct是support
set中类属于c的样本。
原型:对于分类问题,原型网络将其看做在语义空间中寻找每一类的原型中心
将类原型 c 作为任务T的上下文信息,将它们编码到分子嵌入中:
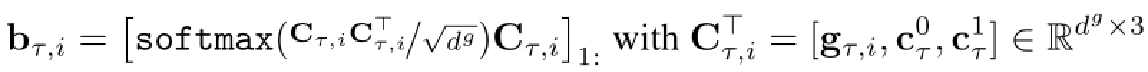
[·]j提取xT,i的第j行向量, C把使用GNN得到的分子嵌入、label为0的类原型 和 label为1的类原型拼起来了。
【其中,bT,i的计算使用了sacled dot-product attention:】
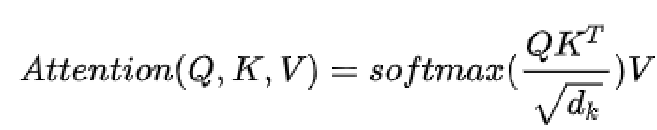
这里bT,i使用缩放点积自注意计算,这样每个gT,i可以以维度方式与类原型进行比较。
缩放点积自注意力:首先对Q和K做点积attention,找到输入之间的相互关系;然后对点积进行尺度变换,也就是缩放点积,其中√d_k是一个缩放因子;使用softmax得到权重然后再与value相乘得到最终值。
缩放点积作用:Softmax函数对非常大的输入值会很敏感。这会导致梯度消失,并减慢学习速度,甚至使其完全停止。由于点积的平均值随着嵌入向量维度 k 的增长而增长,所以将点积的值减小一点有助于防止softmax函数的输入变得过大。让梯度更加平和。关于QKV和注意力机制:你有一个问题Q,然后去搜索引擎里面搜,搜索引擎里面有好多文章,每个标题K对应一篇文章V,然后 搜索引擎用 问题Q 和 标题K 进行匹配,计算相关度(QK —>attention值),然后用不同相关度的 文章V 来表示 问题Q。用这些相关度将检索的文章V做一个加权和,就得到了一个新的Q’,这个Q’融合了相关性强的文章V更多信息,而融合了相关性弱的文章V较少的信息。这就是注意力机制,注意力度不同,重点关注(权值大)与你想要的东西相关性强的部分,稍微关注(权值小)相关性弱的部分。正常的Self-Attention,QKV的输入都来自Decoder上一层的输出。
最后获取 性质感知分子嵌入:
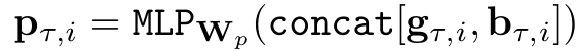
将 GNN得到的分子嵌入g 和 刚刚得到的b 拼接起来 输入到Wp参数化的 多层感知器中,即可得到性质感知分子嵌入pT,i。这种上下文化的属性感知pT,i,可以更好地预测目标属性。
2.3 Adaptive Relation Graph Among Molecules
除了相关的子结构外,分子间的关系也随着性质的变化而变化,共享性质的两个分子 在另一个性质上 可能彼此不同。
因此,本文进一步提出了一个 自适应 关系图 学习模块 来 捕获和利用 分子间的这种 属性感知关系图,使得有限的标签 可以在 相似的分子之间 有效地传播。在这个关系图学习模块中,我们交替地估计分子间关系图的邻接矩阵,并在学习的关系图上细化分子嵌入。
2.3.1 step1:计算分子之间的相似性
GT(t)表示分子之间的关系图;VT表示图中的节点(即分子),包括query set和support set中的分子;AT(t)表示图的邻接矩阵,邻接矩阵中的值 表示两个分子之间的相似性。
首先使用当前的分子嵌入h(t)来估计邻接矩阵AT(t),矩阵中第(i,j)个元素的计算公式为:
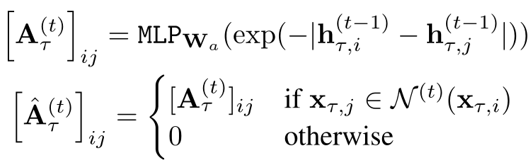
最后,A尖 中的值被归一化为 0~1 之间的范围,这是通过在每一行A上应用 softmax 函数来完成的。
h:分子嵌入。(属性感知分子嵌入之间的相似性揭示了它们在当前属性预测任务下的关系,故令h0=pT,i。)
公式A:e的(两个分子的embedding之差)次方输入到MLP中,输出两个分子之间的关系值,此时参数为Wa。
公式A尖:然而,一个query molecular在图G的2-way K-shot 任务中只有K个真正的邻居 。对于二元分类,尤其是当每个类别中只有一个标记分子时,在相反的类别中选择错误的邻居会严重降低分子嵌入的质量。为了避免错误邻居的干扰,我们进一步将
G(t) 简化为 K近邻 (KNN) 图,其中 K 设置为与 Support set 中每类的标记分子数量完全相同。将领接矩阵A中前K个最大值的 索引 记录在N(t) 中。
2.3.2 step2:计算所有节点(分子)的嵌入
在邻接矩阵AT(t)上,相对于其他节点嵌入,共同调整每个节点嵌入。HT(t)表示所有节点嵌入,其中,Wr是一个可学习的参数。在T次迭代之后,作者将h作为最终的分子xT,i的最终嵌入,A作为最终优化的关系图。
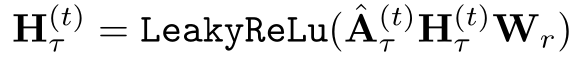
在H中,一行代表一个分子的嵌入
2.3.3 step3:预测分子性质
使用邻居对齐正则化器来惩罚关系图中错误邻居的选择
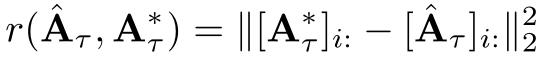
其中:
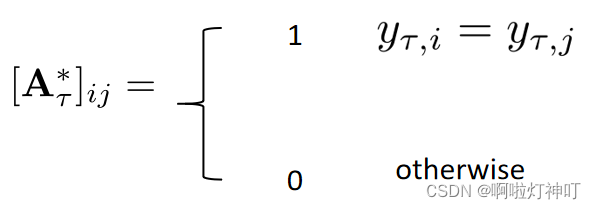
A*是用真实标签计算的,两个分子标签值相等就为1,不等就为0(也就是有关系就是1,没关系就是0)
最后预测类别,由以下公式计算,Wc是可学习的参数
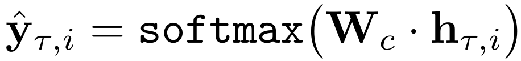
Train loss的计算为:
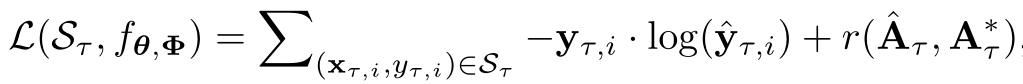
2.4 Algorithm

3. 实验
3.1 实验方法
本文采用基于梯度的元学习策略:从一组元训练任务中学习一个良好的初始化参数,该任务作为每个任务Tτ的起点。基于这种一般策略,有选择地更新任务中的参数,以鼓励模型分别捕获通用和属性感知信息。详细地说,本文保持θ={Wg,Wa,Wr}固定,同时在每个task中微调Φ={Wp,Wc}。通过进行一些梯度下降更新获得Φτ。
3.2 数据集

SIDER是是已上市药物和药物不良反应 (ADR) 的数据库,包含1427种药物和27种副作用。
Tox21是有关化合物毒性的数据库,有7831个分子,12个毒性测量值。
MUV:用于虚拟筛选,包含17种虚拟筛选设计挑战方法
ToxCast是美国环保署(EPA)开始于2007年的一项多年毒理学预测研究项目。ToxCast是利用 快速、标准、自动测试等高通量预筛方法 来识别 潜在的有毒化合物 并对其进行优先排序,它能显著提高筛选的效率,减少常规动物毒性测试的需求量。
3.3 Baseline
(1)没有预训练的基于图的分子编码器的FSL方法:Siamese,ProtoNet,MAML,TPN,EGNN,IterRefLSTM
(2)预训练图的分子编码器方法:Pre-GNN,Meta-MGNN,Pre-PAR
Siamese:孪生网络
ProtoNet:对图像领域的Few-Shot/Zero-Shot任务,应用设计简单的原型网络方法,在通用数据集上达到了state-of-the-art效果
MAML:提出了一种基于梯度下降,学习模型参数的元学习方法,可以学到一组很好的初始化参数
TPN:是在meta-learning的框架中加入传导机制,也就是标签传播来应对少数据的问题。提出Transductive Propagation Network (TPN),对特征嵌入参数和图构建参数进行联合学习。
EGNN:通过图网络进行信息传播,学出边的信息,也就是节点间相似性和相异性
IterRefLSTM:迭代细化LSTM,允许在小分子空间上学习有意义的距离度量
Pre-GNN:同时在图级与节点级对GNN进行预训练,使GNN可以同时学习到图的局部和全局信息
Meta-MGNN:使用meta-learning来学习良好初始化的模型参数,融合了self-supervised module 和 self-attentive module,进一步提高模型性能
Pre-PAR:在本文模型的基础上使用了Pre-GNN
3.4 评估指标
ROC-AUC
3.5 实验结果

①分子图是用RDKit构建的,基于图形的分子编码器选用的是GIN
②最后一列空的值:Siamese、IterRefLSTM 缺少代码,之前没有在 ToxCast 上进行评估,而 Meta-MGNN 内存不足,因为它在meta-training时权衡所有任务中每个任务的贡献
③可以看出,在没有预训练、基于图的few shot learning时,本文PAR效果最好;在有预训练时,本文的Pre-PAR还是最好的。
④学习关系图(即 GNN、TPN、EGNN)的 FSL 方法比经典的 ProtoNet 和 MAML 获得更好的性能。
4. 结论
本文为分子性质预测提出了一种性质感知关系网络(PAR),本文为分子性质预测提出了一种性质感知关系网络(PAR),它与现有的基于图的分子编码器兼容,并进一步具有 获得 属性感知分子嵌入 和 自适应建模分子关系图 的能力。主要工作如下:
①提出了一个属性感知嵌入函数,该函数根据任务的上下文信息共同适应每个分子嵌入,进一步映射到与目标属性相关的子结构感知空间。
②提出了一个自适应关系图学习模块来联合估计分子关系图,并针对目标属性细化分子嵌入,这样有限的标签可以在相似的分子之间有效地传播。
③提出了一种元学习策略来有选择地更新每个任务中的参数(不更新θ更新φ),这有助于捕获不同任务的共享知识。
④在真实的分子性质预测数据集上进行实验,结果表明,PAR 始终优于其他算法。进一步的模型分析表明,PAR 可以正确获得属性感知的分子嵌入和模型分子关系图。