CITE-seq分析 与 scRNA-seq数据分析,两者在数据类型、分析流程、技术重点和结果解读上既有继承又有拓展。
🧬 一、基础定义
| 项目 | scRNA-seq | CITE-seq |
|---|---|---|
| 全称 | Single-cell RNA sequencing | Cellular Indexing of Transcriptomes and Epitopes by Sequencing |
| 类型 | 单模态:只测基因表达 | 多模态:RNA + 表面蛋白(ADT) |
| 数据来源 | mRNA(转录组) | mRNA + 抗体条形码(表面蛋白) |
🧪 二、数据结构对比
✅ scRNA-seq 数据结构:
输入数据矩阵:细胞 × 基因
通常是一个稀疏的 UMI 计数矩阵
✅ CITE-seq 数据结构:
双模态数据:
RNA矩阵:细胞 × 基因(与scRNA-seq相同)
ADT矩阵:细胞 × 抗体标签(即细胞表面蛋白表达)
📌 常用术语:
RNA assay:mRNA表达量(如 CD3E、ACTB 等)
ADT assay:抗体条形码标记的蛋白丰度(如 CD3、CD4、CD8 等)
🔬 三、分析流程对比(Seurat为例)
| 步骤 | scRNA-seq分析 | CITE-seq分析 |
|---|---|---|
| 1️⃣ 预处理 | 数据质控(过滤低质量细胞) | RNA & ADT双通道同时质控 |
| 2️⃣ 归一化 | SCTransform 或 LogNormalize | 分别归一化 RNA 和 ADT(如 CLR) |
| 3️⃣ 降维 | PCA → UMAP/t-SNE | RNA主导降维,也可融合ADT |
| 4️⃣ 聚类 | 基于RNA建图(邻接图、Louvain) | 可融合RNA+ADT建联合图 |
| 5️⃣ 差异分析 | 查找 marker genes | 可同时找 marker genes + marker proteins |
| 6️⃣ 细胞注释 | 基于RNA表达 + 文献比对 | 结合表面蛋白(更准) |
🧠 四、CITE-seq 的优势与应用
✅ 优势:
增强细胞分型准确性(特别是免疫细胞)
蛋白表达比RNA更稳定 → 更少dropout
可识别RNA表达模糊但蛋白分型清晰的细胞群
📌 应用场景:
肿瘤免疫微环境研究
T细胞亚群识别(如 naive vs memory)
多模态联合建模(TotalVI, Seurat v4, MOFA+ 等)
🧰 工具生态对比
| 工具 | 支持scRNA-seq | 支持CITE-seq |
|---|---|---|
| Seurat v3/v4 | ✅ | ✅(多模态支持) |
| Scanpy | ✅ | ✅(通过 muon 或手动处理) |
| TotalVI | ❌ | ✅(专为RNA + ADT建模设计) |
| MOFA+ | ✅ | ✅(任意模态组合) |
| SingleR / Garnett | ✅ | 部分支持 |
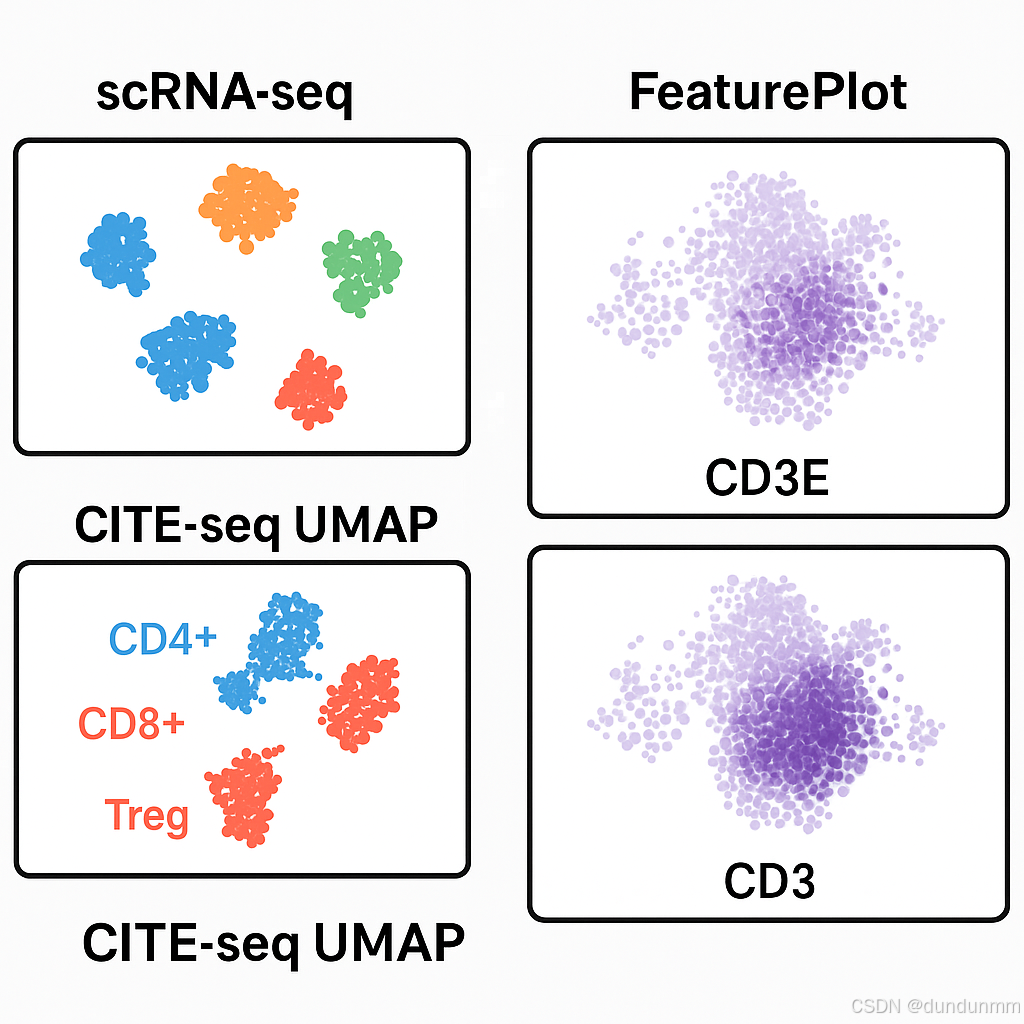
🔎 结果示例图(假想):
scRNA-seq UMAP:细胞群按RNA聚类,可能存在“模糊边界”;
CITE-seq UMAP:更清晰地分出 CD4+ / CD8+ / Treg 等亚型;
FeaturePlot:CD3E(RNA) vs CD3(蛋白)对照,更准确识别细胞类型。
✍️ 总结一句话:
CITE-seq 分析是对传统 scRNA-seq 分析的功能增强,融合了蛋白模态信息,使细胞类型判别更加精细准确,尤其在免疫系统研究中具有重要优势。