问题背景:
直接使用标准图谱和相应的mni标准分区是可以的。
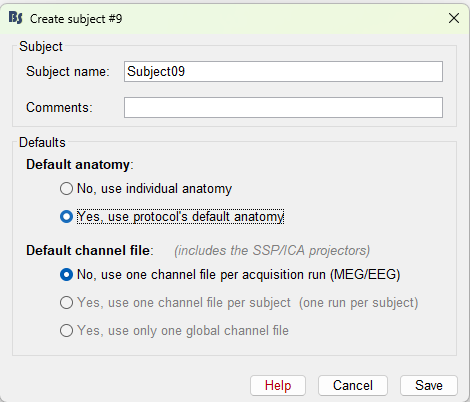
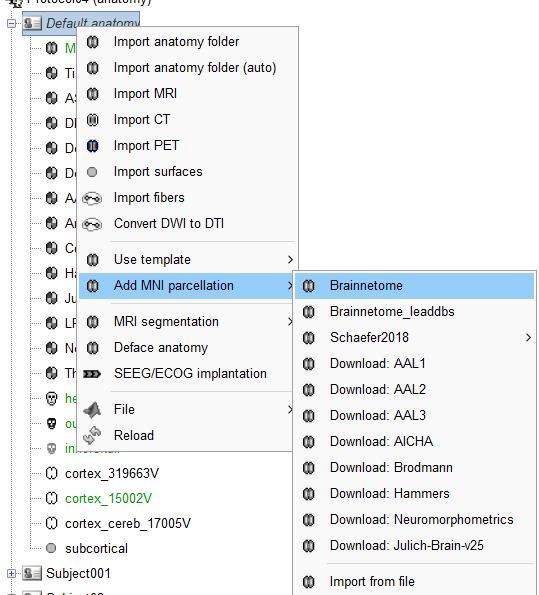
在计算compute
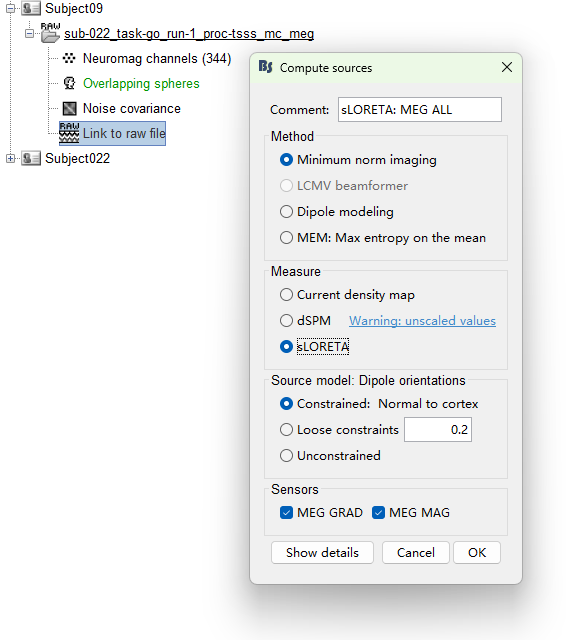
直接使用体积模版进行匹配的脑图谱是不准确的。体积匹配就是,通过上面导入MNI parcellation。这是因为brainnetome采用的需要进行一些变换,才能映射到标准图谱上。但是通过下面的图谱对齐处理,我们可以看到这个显然要平滑一些。
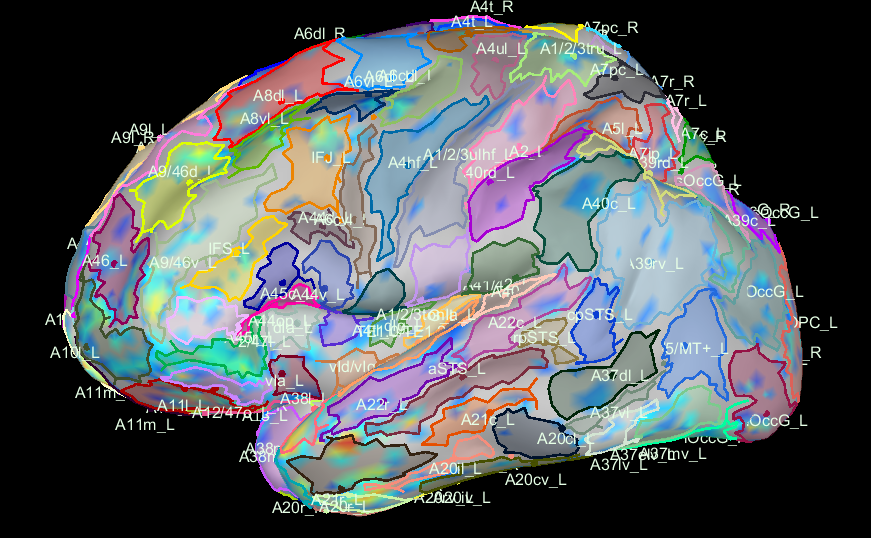
下面这个是使用默认模版直接导入的。
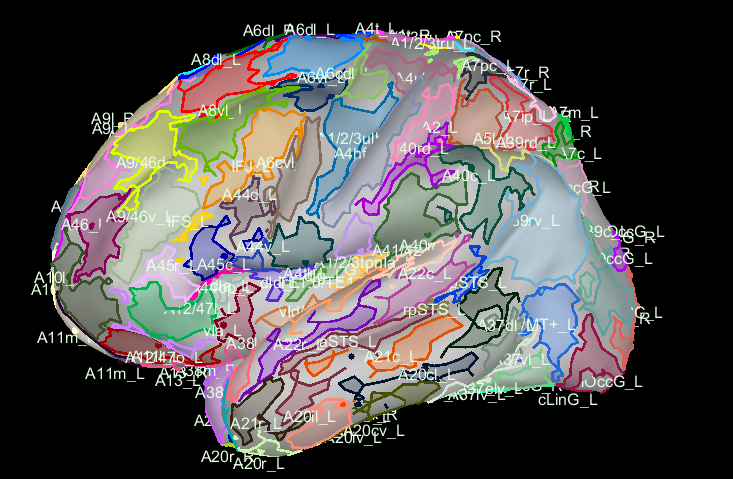
因此下面进行上面这个效果实现的方法说明:
以brainnetome为例。
图谱下载的链接
您要使用
BN_Atlas_246_1mm.nii.gz
BN_Atlas_246_LUT.txt
进行FreeSurfer的被试对齐处理,这通常涉及将个体大脑对齐到标准atlas空间。以下是具体的操作步骤:
1. 准备工作
首先确保您有以下文件:
BN_Atlas_246_1mm.nii.gz(Brainnetome Atlas的NIfTI格式文件)BN_Atlas_246_LUT.txt(查找表文件)- 您的被试数据(通常是T1加权结构像)
2. FreeSurfer环境设置
# 设置FreeSurfer环境变量
export FREESURFER_HOME=/path/to/freesurfer
export SUBJECTS_DIR=/path/to/your/subjects
source $FREESURFER_HOME/SetUpFreeSurfer.sh
3. 被试预处理(如果还没完成)
# 对被试进行完整的FreeSurfer处理
recon-all -i /path/to/subject/T1.nii.gz -subject subjectID -all
4. Atlas对齐操作
方法一:使用mri_vol2vol进行配准
# 将atlas对齐到被试的原生空间
mri_vol2vol --mov BN_Atlas_246_1mm.nii.gz \
--targ $SUBJECTS_DIR/subjectID/mri/T1.mgz \
--regheader \
--o BN_Atlas_in_subject_space.nii.gz \
--nearest
# 或者将被试对齐到atlas空间
mri_vol2vol --mov $SUBJECTS_DIR/subjectID/mri/T1.mgz \
--targ BN_Atlas_246_1mm.nii.gz \
--regheader \
--o subject_in_atlas_space.nii.gz
方法二:使用mri_convert和mri_label2vol
# 转换atlas到FreeSurfer格式
mri_convert BN_Atlas_246_1mm.nii.gz BN_Atlas_246_1mm.mgz
# 创建配准矩阵(如果需要精确配准)
mri_coreg --mov $SUBJECTS_DIR/subjectID/mri/T1.mgz \
--targ BN_Atlas_246_1mm.mgz \
--reg subject_to_atlas.lta
# 应用配准变换
mri_vol2vol --mov BN_Atlas_246_1mm.mgz \
--targ $SUBJECTS_DIR/subjectID/mri/T1.mgz \
--lta subject_to_atlas.lta \
--o BN_Atlas_registered.mgz \
--nearest
5. 验证对齐效果
# 使用freeview查看对齐效果
freeview $SUBJECTS_DIR/subjectID/mri/T1.mgz \
BN_Atlas_registered.mgz:colormap=lut:lut=BN_Atlas_246_LUT.txt
6. ROI提取(可选)
# 提取特定ROI的平均信号
mri_segstats --seg BN_Atlas_registered.mgz \
--ctab BN_Atlas_246_LUT.txt \
--i $SUBJECTS_DIR/subjectID/mri/T1.mgz \
--sum roi_stats.txt
注意事项
- 坐标系统:确保atlas和被试数据使用相同的坐标系统(通常是RAS)
- 分辨率匹配:如果分辨率不匹配,可能需要重采样
- 插值方法:对于标签数据使用
--nearest插值,避免产生新的标签值 - 质量控制:务必通过可视化检查对齐质量
这个流程应该能帮您完成BN Atlas与FreeSurfer被试数据的对齐。如果遇到特定问题,请告诉我详细的错误信息。
进行图谱的功能网络映射
# 创建功能网络映射函数
create_network_mapping() {
local subject_id=$1
local output_dir="$SUBJECTS_DIR/$subject_id/brainnetome"
cat > "$output_dir/functional_networks.txt" << 'EOF'
# Brainnetome 246区域功能网络映射
# 格式: ROI_ID Network_Name Network_ID
# 基于Brainnetome Atlas的8个主要功能网络
# Visual Network (VIS) - 视觉网络
1 Visual 1
2 Visual 1
3 Visual 1
4 Visual 1
17 Visual 1
18 Visual 1
19 Visual 1
20 Visual 1
# Somatomotor Network (SMN) - 躯体运动网络
5 Somatomotor 2
6 Somatomotor 2
7 Somatomotor 2
8 Somatomotor 2
21 Somatomotor 2
22 Somatomotor 2
23 Somatomotor 2
24 Somatomotor 2
57 Somatomotor 2
58 Somatomotor 2
59 Somatomotor 2
60 Somatomotor 2
61 Somatomotor 2
62 Somatomotor 2
63 Somatomotor 2
64 Somatomotor 2
# Dorsal Attention Network (DAN) - 背侧注意网络
9 DorsalAttention 3
10 DorsalAttention 3
25 DorsalAttention 3
26 DorsalAttention 3
73 DorsalAttention 3
74 DorsalAttention 3
75 DorsalAttention 3
76 DorsalAttention 3
# Ventral Attention Network (VAN) - 腹侧注意网络
45 VentralAttention 4
46 VentralAttention 4
47 VentralAttention 4
48 VentralAttention 4
65 VentralAttention 4
66 VentralAttention 4
# Limbic Network (LIM) - 边缘系统网络
89 Limbic 5
90 Limbic 5
91 Limbic 5
92 Limbic 5
93 Limbic 5
94 Limbic 5
95 Limbic 5
96 Limbic 5
211 Limbic 5
212 Limbic 5
213 Limbic 5
214 Limbic 5
215 Limbic 5
216 Limbic 5
# Frontoparietal Network (FPN) - 额顶网络
11 Frontoparietal 6
12 Frontoparietal 6
27 Frontoparietal 6
28 Frontoparietal 6
67 Frontoparietal 6
68 Frontoparietal 6
69 Frontoparietal 6
70 Frontoparietal 6
71 Frontoparietal 6
72 Frontoparietal 6
# Default Mode Network (DMN) - 默认模式网络
77 DefaultMode 7
78 DefaultMode 7
79 DefaultMode 7
80 DefaultMode 7
81 DefaultMode 7
82 DefaultMode 7
83 DefaultMode 7
84 DefaultMode 7
85 DefaultMode 7
86 DefaultMode 7
87 DefaultMode 7
88 DefaultMode 7
# Subcortical Network (SUB) - 皮层下网络
217 Subcortical 8
218 Subcortical 8
219 Subcortical 8
220 Subcortical 8
221 Subcortical 8
222 Subcortical 8
223 Subcortical 8
224 Subcortical 8
225 Subcortical 8
226 Subcortical 8
227 Subcortical 8
228 Subcortical 8
229 Subcortical 8
230 Subcortical 8
231 Subcortical 8
232 Subcortical 8
233 Subcortical 8
234 Subcortical 8
235 Subcortical 8
236 Subcortical 8
237 Subcortical 8
238 Subcortical 8
239 Subcortical 8
240 Subcortical 8
241 Subcortical 8
242 Subcortical 8
243 Subcortical 8
244 Subcortical 8
245 Subcortical 8
246 Subcortical 8
EOF
}
此处把所有的批处理文件放在这里,使用freesurfer来执行。可以生成可导入brainstorm的包含,brainnetome图谱和对应的功能网络分区的scouts。
可以自行修改。
#!/bin/bash
# FreeSurfer + Brainnetome Atlas处理脚本 - 用于MEG源分析
# 用法:./process_deformed_templates_brainnetome.sh
# 设置FreeSurfer环境
#export FREESURFER_HOME=/path/to/freesurfer # 修改为您的FreeSurfer路径
#source $FREESURFER_HOME/SetUpFreeSurfer.sh
export SUBJECTS_DIR=$(pwd)/FS_output # 输出目录
# 创建输出目录
mkdir -p $SUBJECTS_DIR
# 检查Brainnetome图谱文件
BRAINNETOME_ATLAS="$SUBJECTS_DIR/BN_Atlas_246_1mm.nii.gz"
BRAINNETOME_LUT="$SUBJECTS_DIR/BN_Atlas_246_LUT.txt"
if [ ! -f "$BRAINNETOME_ATLAS" ]; then
echo "错误: 找不到Brainnetome图谱文件: $BRAINNETOME_ATLAS"
exit 1
fi
if [ ! -f "$BRAINNETOME_LUT" ]; then
echo "错误: 找不到Brainnetome LUT文件: $BRAINNETOME_LUT"
exit 1
fi
echo "找到Brainnetome图谱文件:"
echo " 图谱: $BRAINNETOME_ATLAS"
echo " LUT: $BRAINNETOME_LUT"
# 线程数设置
OPENMP_THREADS=8
# 创建功能网络映射函数
create_network_mapping() {
local subject_id=$1
local output_dir="$SUBJECTS_DIR/$subject_id/brainnetome"
cat > "$output_dir/functional_networks.txt" << 'EOF'
# Brainnetome 246区域功能网络映射
# 格式: ROI_ID Network_Name Network_ID
# 基于Brainnetome Atlas的8个主要功能网络
# Visual Network (VIS) - 视觉网络
1 Visual 1
2 Visual 1
3 Visual 1
4 Visual 1
17 Visual 1
18 Visual 1
19 Visual 1
20 Visual 1
# Somatomotor Network (SMN) - 躯体运动网络
5 Somatomotor 2
6 Somatomotor 2
7 Somatomotor 2
8 Somatomotor 2
21 Somatomotor 2
22 Somatomotor 2
23 Somatomotor 2
24 Somatomotor 2
57 Somatomotor 2
58 Somatomotor 2
59 Somatomotor 2
60 Somatomotor 2
61 Somatomotor 2
62 Somatomotor 2
63 Somatomotor 2
64 Somatomotor 2
# Dorsal Attention Network (DAN) - 背侧注意网络
9 DorsalAttention 3
10 DorsalAttention 3
25 DorsalAttention 3
26 DorsalAttention 3
73 DorsalAttention 3
74 DorsalAttention 3
75 DorsalAttention 3
76 DorsalAttention 3
# Ventral Attention Network (VAN) - 腹侧注意网络
45 VentralAttention 4
46 VentralAttention 4
47 VentralAttention 4
48 VentralAttention 4
65 VentralAttention 4
66 VentralAttention 4
# Limbic Network (LIM) - 边缘系统网络
89 Limbic 5
90 Limbic 5
91 Limbic 5
92 Limbic 5
93 Limbic 5
94 Limbic 5
95 Limbic 5
96 Limbic 5
211 Limbic 5
212 Limbic 5
213 Limbic 5
214 Limbic 5
215 Limbic 5
216 Limbic 5
# Frontoparietal Network (FPN) - 额顶网络
11 Frontoparietal 6
12 Frontoparietal 6
27 Frontoparietal 6
28 Frontoparietal 6
67 Frontoparietal 6
68 Frontoparietal 6
69 Frontoparietal 6
70 Frontoparietal 6
71 Frontoparietal 6
72 Frontoparietal 6
# Default Mode Network (DMN) - 默认模式网络
77 DefaultMode 7
78 DefaultMode 7
79 DefaultMode 7
80 DefaultMode 7
81 DefaultMode 7
82 DefaultMode 7
83 DefaultMode 7
84 DefaultMode 7
85 DefaultMode 7
86 DefaultMode 7
87 DefaultMode 7
88 DefaultMode 7
# Subcortical Network (SUB) - 皮层下网络
217 Subcortical 8
218 Subcortical 8
219 Subcortical 8
220 Subcortical 8
221 Subcortical 8
222 Subcortical 8
223 Subcortical 8
224 Subcortical 8
225 Subcortical 8
226 Subcortical 8
227 Subcortical 8
228 Subcortical 8
229 Subcortical 8
230 Subcortical 8
231 Subcortical 8
232 Subcortical 8
233 Subcortical 8
234 Subcortical 8
235 Subcortical 8
236 Subcortical 8
237 Subcortical 8
238 Subcortical 8
239 Subcortical 8
240 Subcortical 8
241 Subcortical 8
242 Subcortical 8
243 Subcortical 8
244 Subcortical 8
245 Subcortical 8
246 Subcortical 8
EOF
}
# Brainnetome图谱处理函数
process_brainnetome_atlas() {
local subject_id=$1
local subject_dir="$SUBJECTS_DIR/$subject_id"
local bn_output_dir="$subject_dir/brainnetome"
echo "开始Brainnetome图谱处理: $subject_id"
# 创建brainnetome输出目录
mkdir -p "$bn_output_dir"
# 1. 将Brainnetome图谱配准到被试空间
echo " 配准Brainnetome图谱到被试空间..."
mri_vol2vol --mov "$BRAINNETOME_ATLAS" \
--targ "$subject_dir/mri/T1.mgz" \
--regheader \
--o "$bn_output_dir/BN_Atlas_246_subject_space.mgz" \
--nearest
if [ $? -ne 0 ]; then
echo " 错误: Brainnetome图谱配准失败"
return 1
fi
# 2. 转换为NIFTI格式便于后续分析
echo " 转换为NIFTI格式..."
mri_convert "$bn_output_dir/BN_Atlas_246_subject_space.mgz" \
"$bn_output_dir/BN_Atlas_246_subject_space.nii.gz"
# 3. 生成被试特异的LUT文件
echo " 复制LUT文件..."
cp "$BRAINNETOME_LUT" "$bn_output_dir/BN_Atlas_246_LUT.txt"
# 4. 提取图谱统计信息
echo " 提取图谱统计信息..."
mri_segstats --seg "$bn_output_dir/BN_Atlas_246_subject_space.mgz" \
--ctab "$BRAINNETOME_LUT" \
--sum "$bn_output_dir/brainnetome_stats.txt" \
--pv "$subject_dir/mri/T1.mgz"
# 5. 创建功能网络映射
echo " 创建功能网络映射..."
create_network_mapping "$subject_id"
# 6. 生成每个ROI的mask
echo " 生成ROI masks..."
local roi_dir="$bn_output_dir/rois"
mkdir -p "$roi_dir"
# 从LUT文件读取ROI信息并生成masks
while IFS=$'\t' read -r roi_id roi_name r g b a; do
# 跳过注释行和空行
if [[ $roi_id =~ ^#.*$ ]] || [ -z "$roi_id" ]; then
continue
fi
# 生成单个ROI的mask
mri_binarize --i "$bn_output_dir/BN_Atlas_246_subject_space.mgz" \
--match "$roi_id" \
--o "$roi_dir/ROI_${roi_id}_${roi_name// /_}.mgz" \
> /dev/null 2>&1
done < "$BRAINNETOME_LUT"
# 7. 生成网络级别的masks
echo " 生成功能网络masks..."
local network_dir="$bn_output_dir/networks"
mkdir -p "$network_dir"
# 为每个功能网络创建mask
for network_id in {1..8}; do
case $network_id in
1) network_name="Visual" ;;
2) network_name="Somatomotor" ;;
3) network_name="DorsalAttention" ;;
4) network_name="VentralAttention" ;;
5) network_name="Limbic" ;;
6) network_name="Frontoparietal" ;;
7) network_name="DefaultMode" ;;
8) network_name="Subcortical" ;;
esac
# 提取属于该网络的ROI ID
roi_list=$(awk -v net_id="$network_id" '$3 == net_id {printf "%s ", $1}' "$bn_output_dir/functional_networks.txt")
if [ ! -z "$roi_list" ]; then
# 创建网络mask
mri_binarize --i "$bn_output_dir/BN_Atlas_246_subject_space.mgz" \
--match $roi_list \
--o "$network_dir/Network_${network_id}_${network_name}.mgz"
fi
done
# 8. 生成Brainnetome处理总结
cat > "$bn_output_dir/BRAINNETOME_PROCESSING_SUMMARY.txt" << EOF
Brainnetome Atlas处理总结
被试ID: $subject_id
处理日期: $(date)
生成文件:
1. 配准图谱: BN_Atlas_246_subject_space.mgz/nii.gz
2. LUT文件: BN_Atlas_246_LUT.txt
3. 统计文件: brainnetome_stats.txt
4. 功能网络映射: functional_networks.txt
5. ROI masks: rois/ROI_*.mgz (246个区域)
6. 网络masks: networks/Network_*.mgz (8个功能网络)
功能网络:
1. Visual (视觉网络)
2. Somatomotor (躯体运动网络)
3. DorsalAttention (背侧注意网络)
4. VentralAttention (腹侧注意网络)
5. Limbic (边缘系统网络)
6. Frontoparietal (额顶网络)
7. DefaultMode (默认模式网络)
8. Subcortical (皮层下网络)
使用方法:
- MEG源分析: 使用rois/中的单个ROI masks
- 网络分析: 使用networks/中的网络masks
- 连接组分析: 使用brainnetome_stats.txt中的统计信息
EOF
echo " Brainnetome图谱处理完成"
return 0
}
# 处理当前目录下所有符合条件的NIFTI文件
for input_file in ./*_desc-warpimg_T1w.nii; do
# 提取被试ID (从文件名如sub-001_desc-warpimg_T1w.nii提取sub-001)
subject_id=$(basename "$input_file" | cut -d'_' -f1)
# 检查是否已处理过
if [ -d "$SUBJECTS_DIR/$subject_id" ]; then
# 检查是否已完成所需步骤
if [ -f "$SUBJECTS_DIR/$subject_id/COMPLETED_MEG_PROCESSING" ] && \
[ -f "$SUBJECTS_DIR/$subject_id/COMPLETED_BRAINNETOME_PROCESSING" ]; then
echo "跳过已处理被试: $subject_id"
continue
else
echo "检测到未完成处理,重新处理被试: $subject_id"
# 保留已完成的FreeSurfer处理,只重新处理Brainnetome部分
if [ ! -f "$SUBJECTS_DIR/$subject_id/COMPLETED_MEG_PROCESSING" ]; then
rm -rf "$SUBJECTS_DIR/$subject_id"
fi
fi
fi
echo "开始处理: $subject_id"
echo "输入文件: $input_file"
# FreeSurfer处理部分
if [ ! -f "$SUBJECTS_DIR/$subject_id/COMPLETED_MEG_PROCESSING" ]; then
# 执行完整的recon-all
echo "执行完整recon-all处理..."
recon-all -s "$subject_id" -i "$input_file" -all -openmp $OPENMP_THREADS
# 检查recon-all是否成功
if [ $? -ne 0 ]; then
echo "recon-all处理失败: $subject_id"
echo "请检查日志: $SUBJECTS_DIR/$subject_id/scripts/recon-all.log"
continue
fi
# 验证关键文件是否生成
echo "验证FreeSurfer文件..."
required_files=(
"$SUBJECTS_DIR/$subject_id/surf/lh.pial"
"$SUBJECTS_DIR/$subject_id/surf/rh.pial"
"$SUBJECTS_DIR/$subject_id/surf/lh.white"
"$SUBJECTS_DIR/$subject_id/surf/rh.white"
"$SUBJECTS_DIR/$subject_id/surf/lh.sphere.reg"
"$SUBJECTS_DIR/$subject_id/surf/rh.sphere.reg"
"$SUBJECTS_DIR/$subject_id/surf/lh.inflated"
"$SUBJECTS_DIR/$subject_id/surf/rh.inflated"
"$SUBJECTS_DIR/$subject_id/mri/T1.mgz"
"$SUBJECTS_DIR/$subject_id/mri/brain.mgz"
"$SUBJECTS_DIR/$subject_id/mri/aseg.mgz"
)
missing_files=()
for file in "${required_files[@]}"; do
if [ ! -f "$file" ]; then
missing_files+=("$file")
fi
done
if [ ${#missing_files[@]} -gt 0 ]; then
echo "警告: 以下FreeSurfer文件缺失:"
printf '%s\n' "${missing_files[@]}"
echo "被试 $subject_id FreeSurfer处理不完整"
continue
fi
# 生成FreeSurfer处理总结
echo "生成FreeSurfer处理总结..."
cat > "$SUBJECTS_DIR/$subject_id/MEG_PROCESSING_SUMMARY.txt" << EOF
被试ID: $subject_id
FreeSurfer处理日期: $(date)
输入文件: $input_file
生成的关键文件(用于Brainstorm):
- 皮层表面: lh.pial, rh.pial, lh.white, rh.white
- 表面配准: lh.sphere.reg, rh.sphere.reg
- 膨胀表面: lh.inflated, rh.inflated
- 解剖信息: mri/T1.mgz, mri/brain.mgz, mri/aseg.mgz
处理阶段:
✓ 完整recon-all处理: 完成
✓ 使用参数: -all -openmp $OPENMP_THREADS
注意: 此数据使用变形的T1图像处理,用于MEG源定位分析
EOF
# 创建FreeSurfer完成标记
touch "$SUBJECTS_DIR/$subject_id/COMPLETED_MEG_PROCESSING"
echo "FreeSurfer处理完成: $subject_id"
else
echo "FreeSurfer已完成,跳过: $subject_id"
fi
# Brainnetome图谱处理部分
if [ ! -f "$SUBJECTS_DIR/$subject_id/COMPLETED_BRAINNETOME_PROCESSING" ]; then
if process_brainnetome_atlas "$subject_id"; then
# 创建Brainnetome完成标记
touch "$SUBJECTS_DIR/$subject_id/COMPLETED_BRAINNETOME_PROCESSING"
echo "Brainnetome处理完成: $subject_id"
else
echo "Brainnetome处理失败: $subject_id"
continue
fi
else
echo "Brainnetome已完成,跳过: $subject_id"
fi
echo "被试 $subject_id 全部处理完成!"
echo "------------------------------------------------"
done
echo ""
echo "所有处理完成!"
echo "输出目录: $SUBJECTS_DIR"
echo ""
echo "每个被试的输出结构:"
echo " $SUBJECTS_DIR/sub-xxx/"
echo " ├── surf/ # FreeSurfer表面文件"
echo " ├── mri/ # FreeSurfer解剖文件"
echo " ├── brainnetome/"
echo " │ ├── BN_Atlas_246_subject_space.mgz/nii.gz # 配准的图谱"
echo " │ ├── BN_Atlas_246_LUT.txt # 标签文件"
echo " │ ├── brainnetome_stats.txt # 统计信息"
echo " │ ├── functional_networks.txt # 功能网络映射"
echo " │ ├── rois/ # 246个ROI masks"
echo " │ └── networks/ # 8个功能网络masks"
echo " ├── MEG_PROCESSING_SUMMARY.txt"
echo " └── BRAINNETOME_PROCESSING_SUMMARY.txt"
echo ""
echo "Brainstorm + MEG源分析使用说明:"
echo "1. FreeSurfer数据导入Brainstorm (指向 $SUBJECTS_DIR/sub-xxx)"
echo "2. ROI分析: 使用 brainnetome/rois/ 中的单个区域masks"
echo "3. 网络分析: 使用 brainnetome/networks/ 中的功能网络masks"
echo "4. 连接组分析: 参考 brainnetome_stats.txt 和 functional_networks.txt"
echo ""
echo "功能网络包括:"
echo " 1. Visual (视觉网络)"
echo " 2. Somatomotor (躯体运动网络)"
echo " 3. DorsalAttention (背侧注意网络)"
echo " 4. VentralAttention (腹侧注意网络)"
echo " 5. Limbic (边缘系统网络)"
echo " 6. Frontoparietal (额顶网络)"
echo " 7. DefaultMode (默认模式网络)"
echo " 8. Subcortical (皮层下网络)"