一直以来对牛津纳米孔测序技术比较好奇,想要测试下他官方的分析软件,无奈之前的软件很封闭,只能买机器才能用,这两天发现可以以游客身份安装使用啦,这里测试一下。
EPI2ME 是 Oxford Nanopore Technologies 提供的一套生物信息学工作流程平台,专为其长读长测序数据设计。它基于 Nextflow 框架,支持容器技术和软件包管理器,适用于本地、集群和云端部署,非常适合需要高扩展性的科研环境。
命令行流程测试
后面提到,图形界面折腾失败,只能退而求其次,上命令行啦!之前就有安装过nextflow的conda版本,是个独立的环境,于是更新了下版本,用起来!同样面临到了docker hub的网络问题,找了个镜像网址解决的。一个样本默认是用4个线程进行minimap2比对的。
测试数据用的这个:https://trace.ncbi.nlm.nih.gov/Traces/?view=run_browser&acc=SRR30050020&display=metadata
文档在这里:https://epi2me.nanoporetech.com/epi2me-docs/workflows/wf-16s/(和github是同步的,https://github.com/epi2me-labs/wf-16s)
# 好久不用,运行流程报错,升级下
mamba update nextflow
# 拉取流程并运行,会下载两个docker镜像
nextflow run epi2me-labs/wf-16s --fastq /mnt/d/epime/data/SRR30050020.fastq.gz --minimap2_by_reference -profile standard
二十多分钟后,就愉快地有了报告,还是网页版本,不错的。有个插曲就是硬件配置不足,一次是修改了WSL的硬件配置,拉满到了全部硬件,就是修改了windows用户目录下的.wslconfig文件。另一个就是报硬件不足,本来以为要失败时想了下,应该是数据一百多M过大,于是抽取了前10万条,终于运行成功啦!
结果查看
报告有两个,一个是流程的运行情况,这个用处不太大,一般是用于优化算力上的流程参数的。
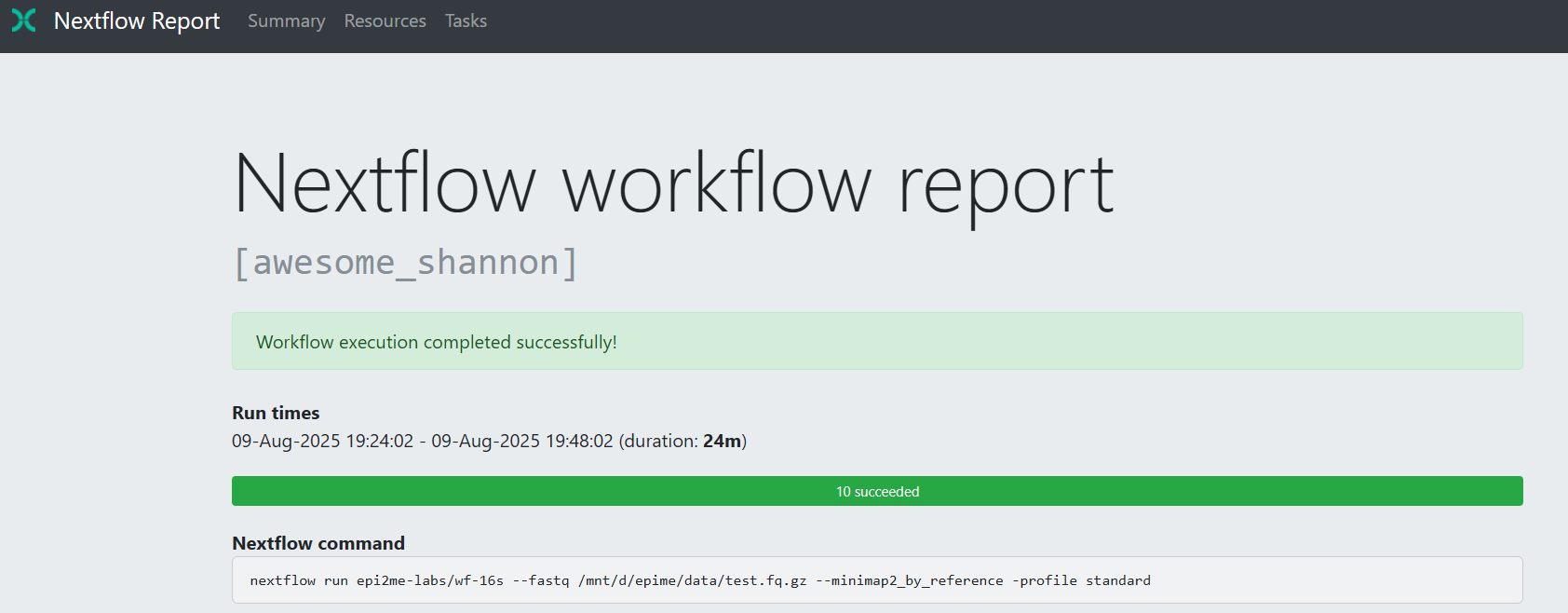
另一个就是我们最关心的结果报告啦,官网也给出了示例版本:https://epi2me.nanoporetech.com/workflows/wf-16s/wf-16s-report.html#Section_b90ae906eb624f53a77553eba1782c6c
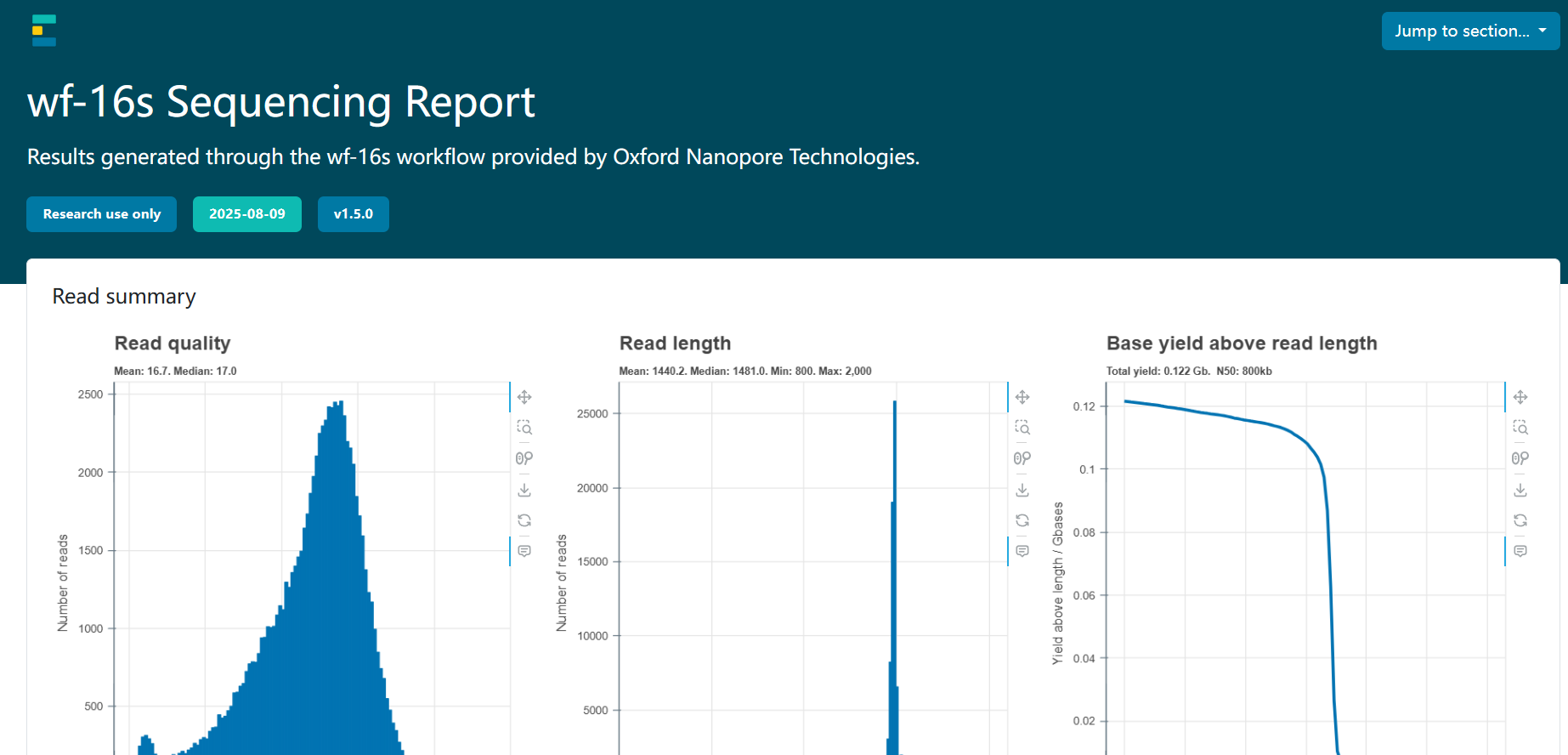
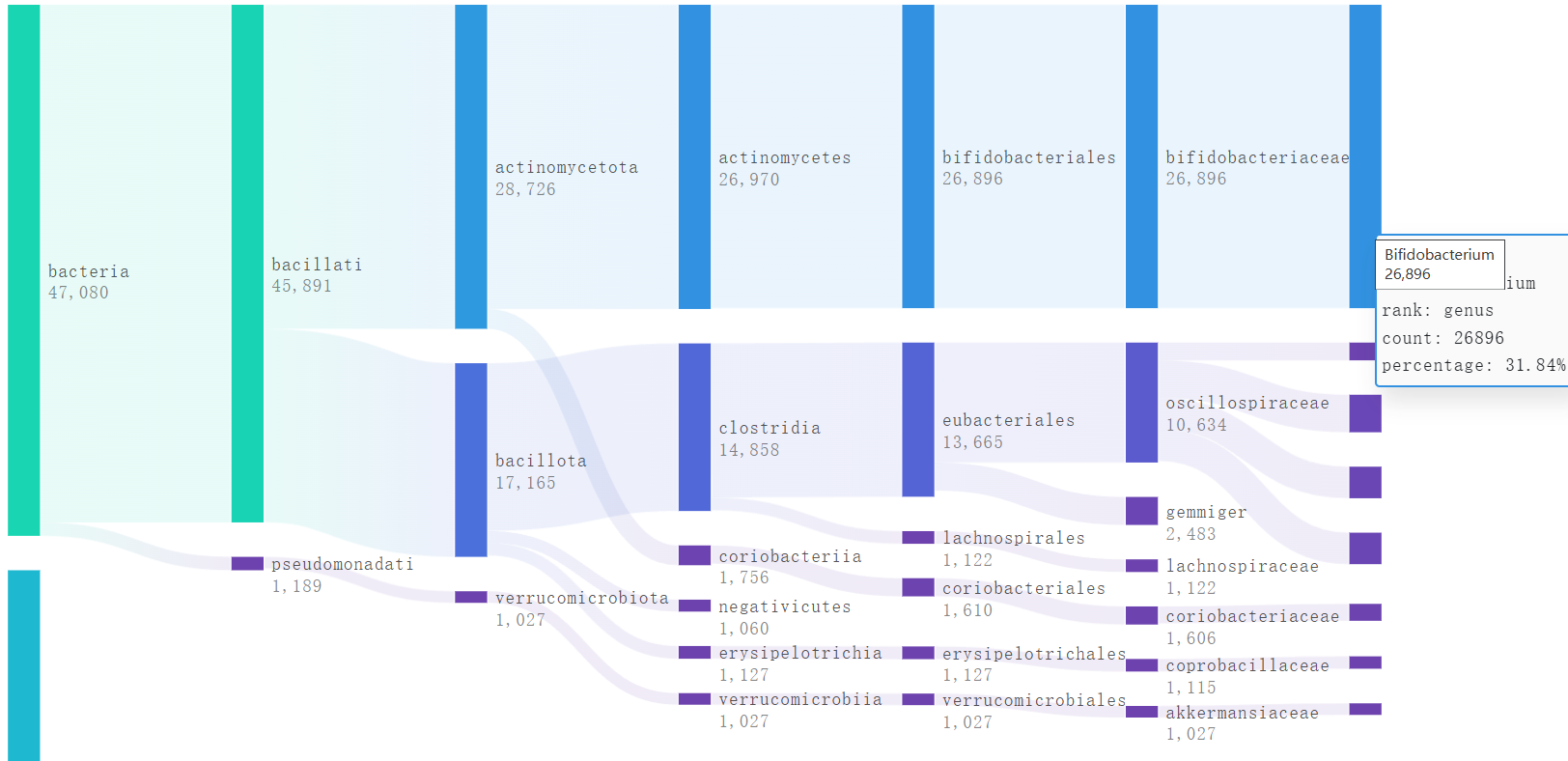
稍微有点失望的是只注释到了属,有的还不到,可能原因是数据是旧的芯片版本或者nanopore对16S的测序还确实不太如人意呀!多样性指数,可视化还是不错的,圆盘,百分比堆积柱状图,桑基图,稀释曲线,优势物种图都是有的。
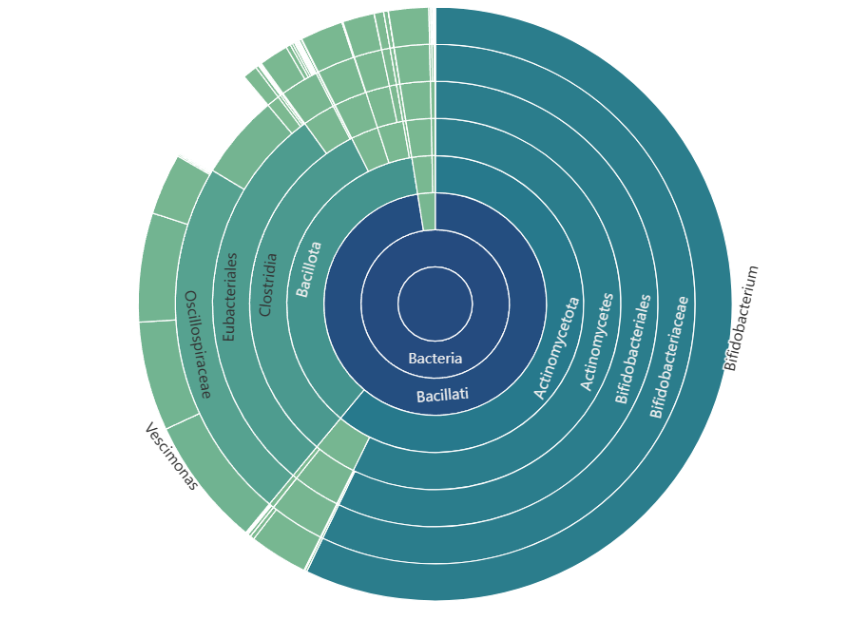
还有个kraken2的流程,没有找到详细的运行参数,没做尝试,相信结果也是类似的啦。初步总结,还是基本满足需求的,特别是对于分析基础相对弱的,有现成的软件工具可用,方便很多。
图形界面的尝试
就直接跟着官网的步骤下载安装啦,网址在这:https://epi2me.nanoporetech.com/downloads/,windows, mac, linux三大平台都是有的。官网说,对于外行来说,安装生物信息学软件可能是出了名的困难。 EPI2ME 桌面应用程序旨在允许任何人随时随地分析任何内容。 简化了 Oxford Nanopore Technologies 测序应用的生物信息学工具和资源的安装和使用。
我是windows版本啦,主要是还是相对来讲性价比最高的平台嘛。开始是需要登陆的,但是现在可以以游客方式使用,更方便了。
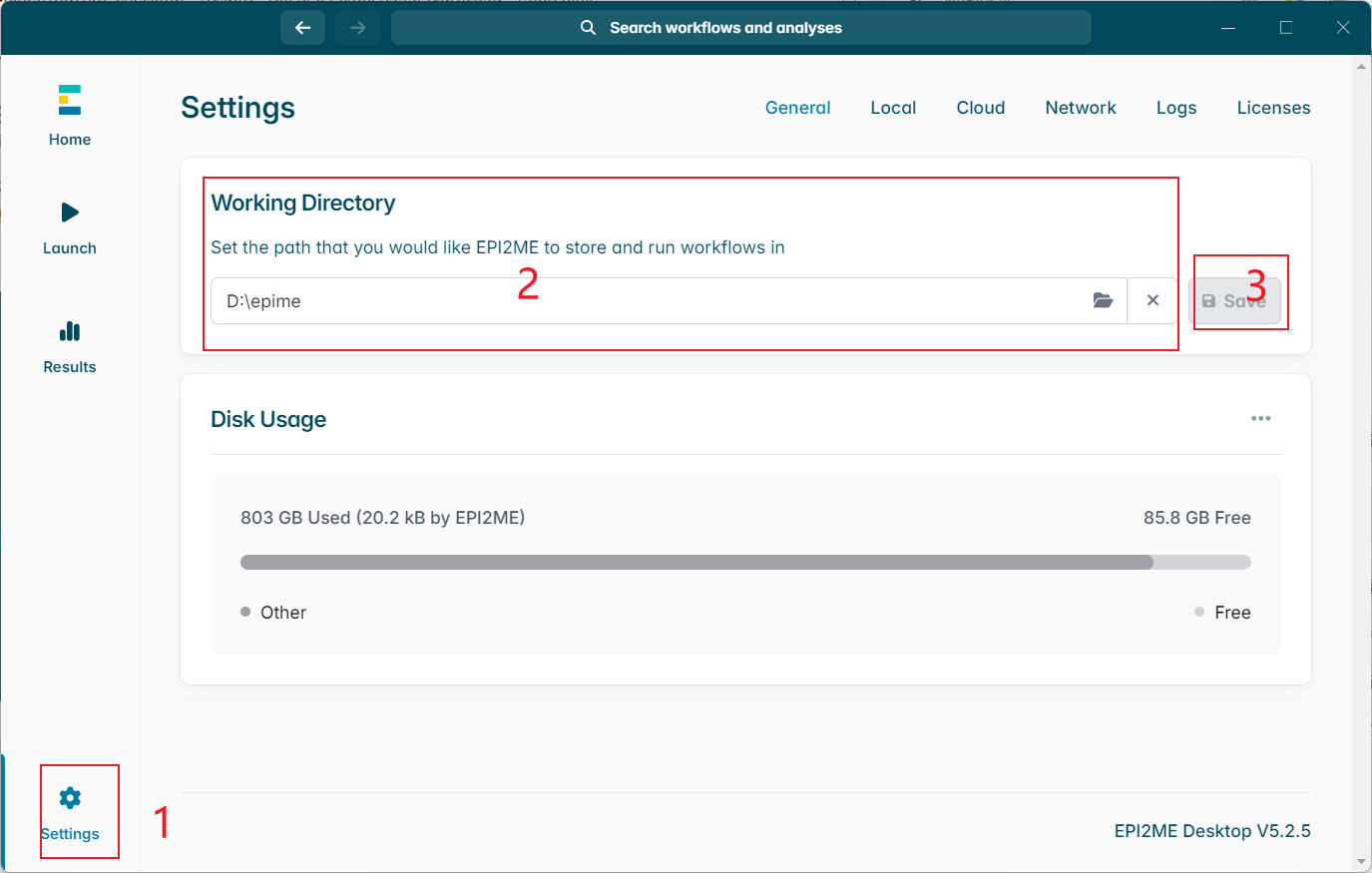
然后就是配置保存目录啦,防止系统盘过小,一下就整爆了,另外依赖的WSL和docker也是可以选择路径的哦。
配置WSL,必须是官方的版本,还要求最新的,是epime专用的,也不知道密码。。。
下面是详细配置步骤:
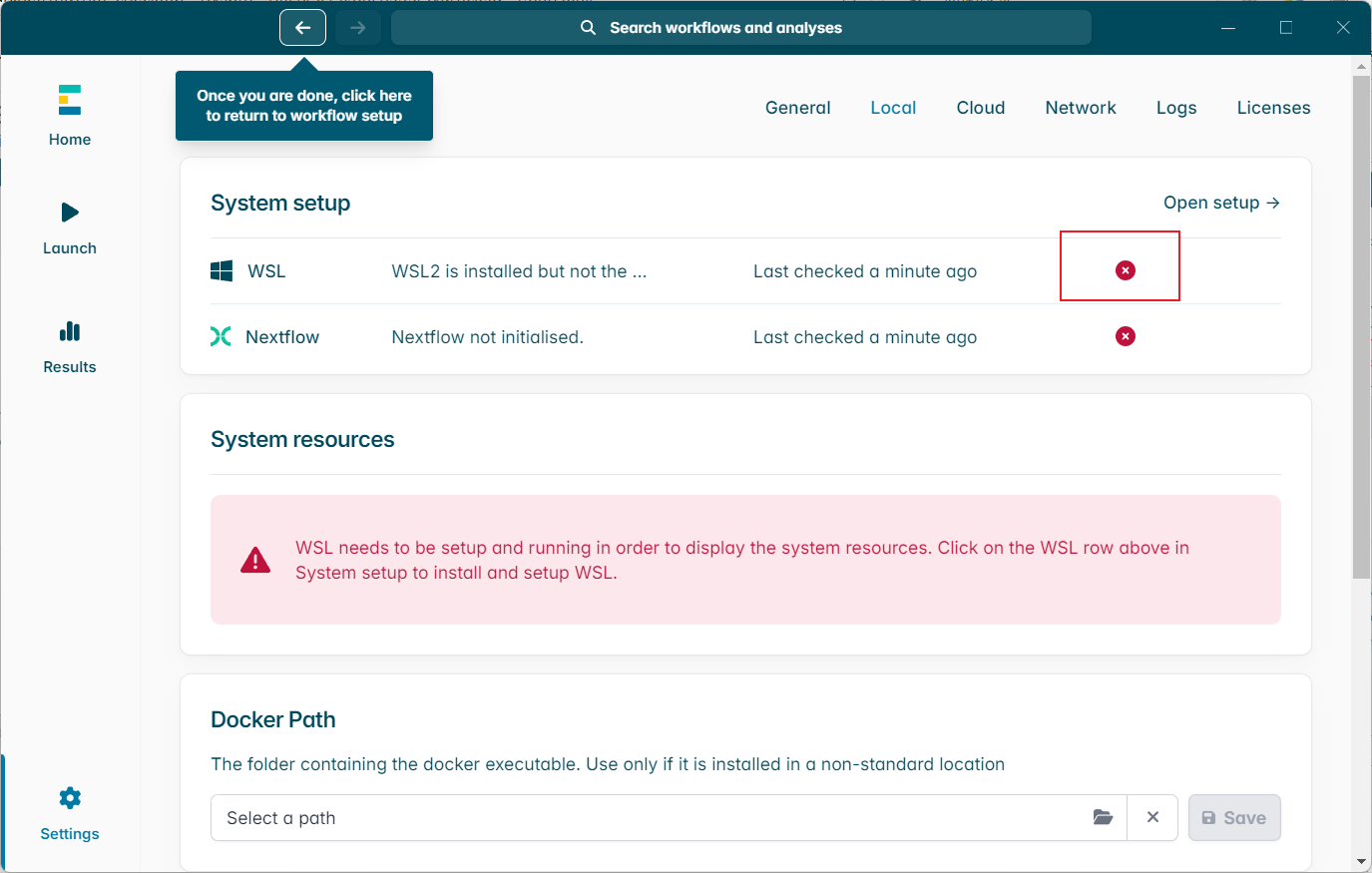
相对傻瓜的,毕竟是图形软件,不得不说ONT做得不错。不过这个折腾还是有点时间久的,毕竟要下载一个相当于小系统镜像。
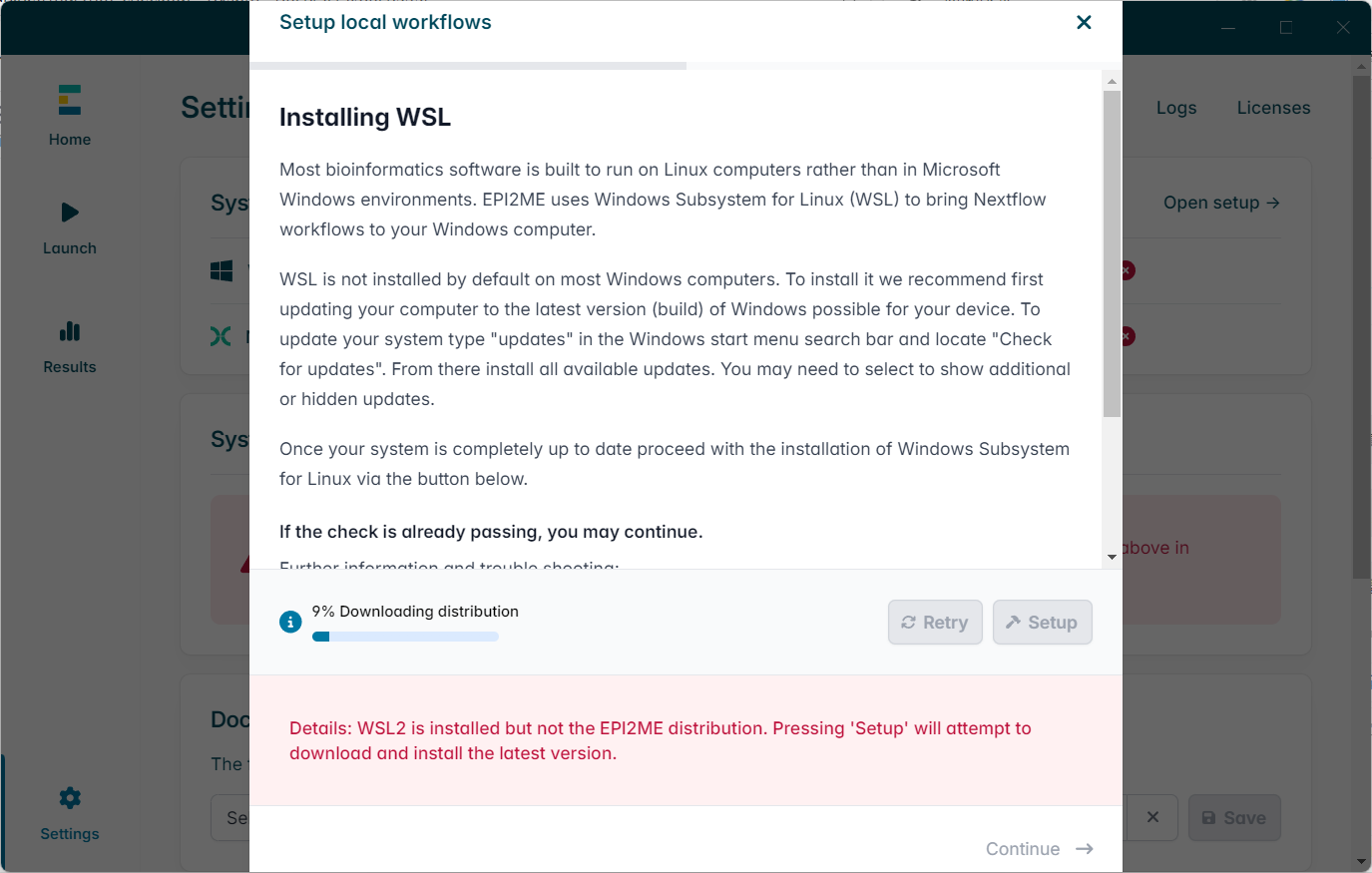
安装完WSL,就是流程的nextflow啦,这个是个应用,更轻松啦就。
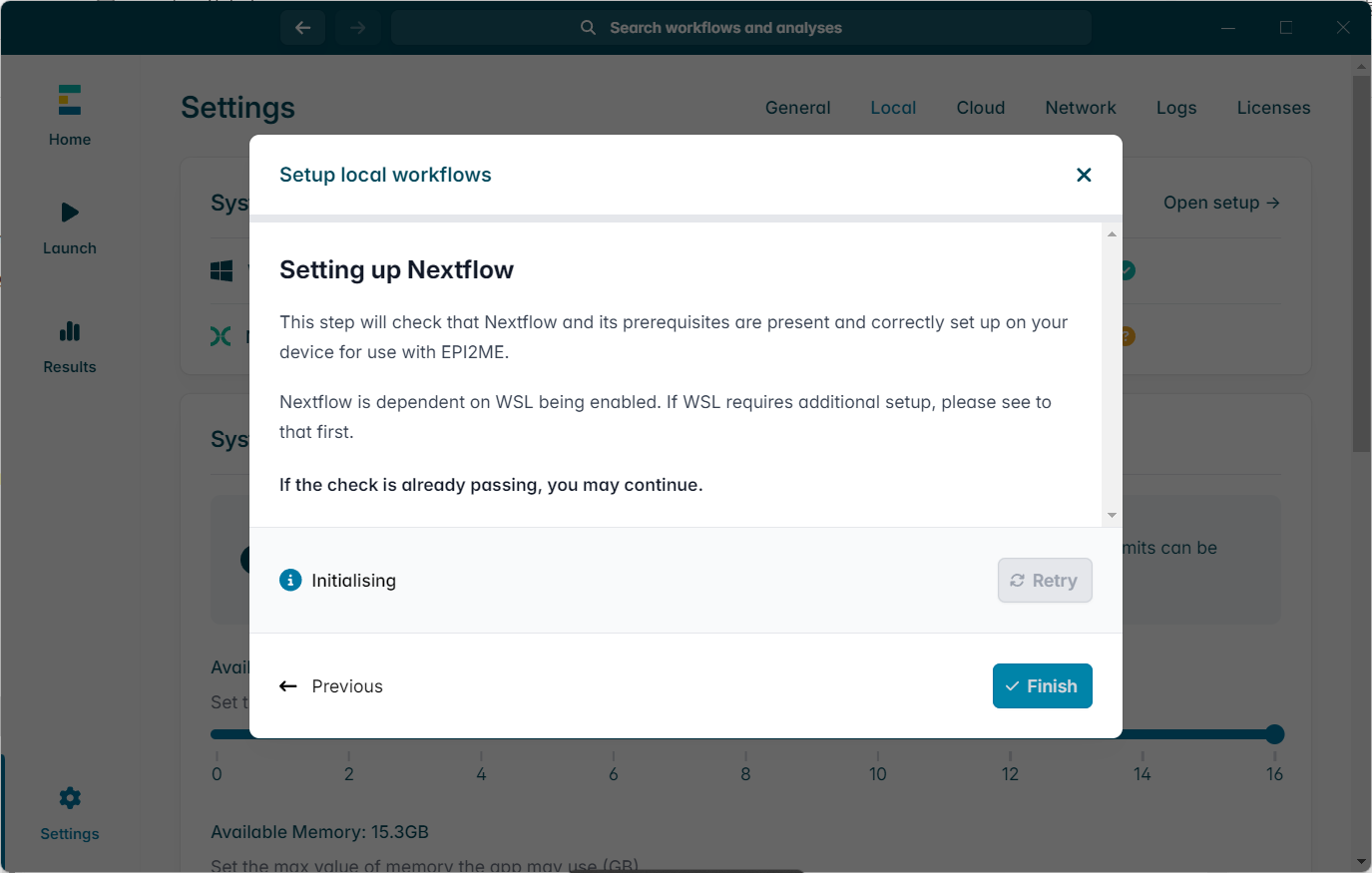
完工,开始配置流程,就卡在这了,原因很简单,就是docker hub连接不上,有途径的帅美可以考虑途径解决啦。
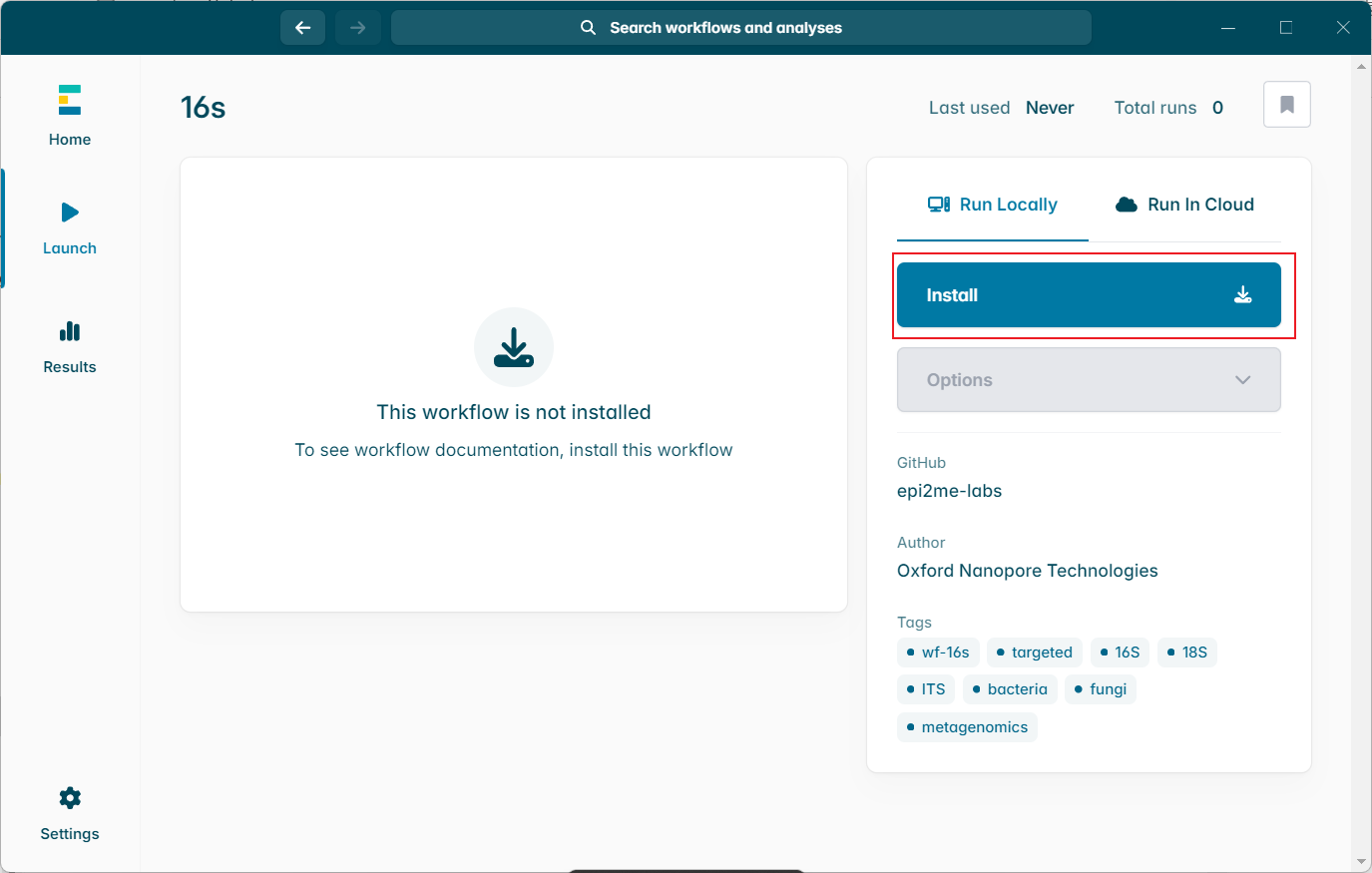
推荐内存32G,新测确实,如果文件超过100M,就会报错RAM不足的。
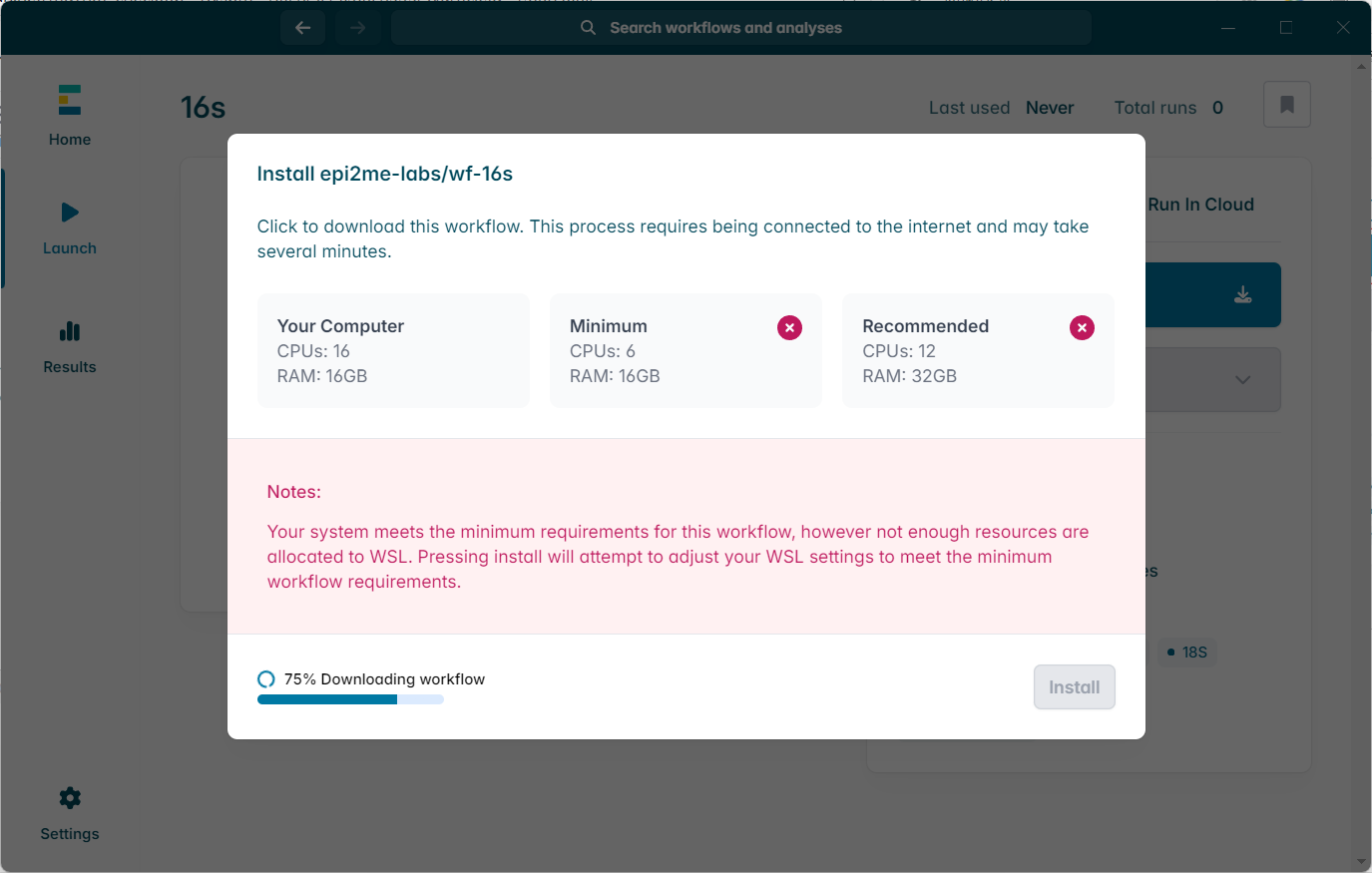
没能解决软件如何配置网络的问题,只能换个思路啦,好在还有命令行的公开,如果网络问题解决,还是很轻松就完成啦!