大家好,这里是专注表观组学十余年,领跑多组学科研服务的易基因。

2022年09月24日,陆军医科大学(第三军医大学)预防医学院毒理学研究所张中豪博士等为第一作者,曹佳教授、刘晋祎教授为通讯作者以" Genome-wide alternation and effect of DNA methylation in the impairments of steroidogenesis and spermatogenesis after PM2.5 exposure"为题在《Environment International》杂志发表研究论文。该研究通过对小鼠睾丸组织的全基因组重亚硫酸盐测序(Whole gene bisulfite sequencing,WGBS)与对应的转录组测序(RNA-seq)联合分析,鉴定出与类固醇生成和精子发生过程相关的关键基因和通路,揭示了PM2.5导致男性生殖障碍的潜在DNA甲基化调控机制。深圳市易基因科技有限公司提供本研究的DNA甲基化测序(WGBS)和转录组测序(RNA-seq)服务。
标题:Genome-wide alternation and effect of DNA methylation in the impairments of steroidogenesis and spermatogenesis after PM2.5 exposure(PM2.5暴露后类固醇生成和精子生成障碍的全基因组DNA甲基化变化和效应)
发表期刊:Environment International
发表日期:2022年09月24日
影响因子:IF 13.352
技术:全基因组重亚硫酸盐测序(WGBS)、转录组测序(RNA-seq)、蛋白质印记
样品:8周龄C57BL/6雄性小鼠,分为过滤空气组(filtered air group ,FA),未过滤空气组(unfiltered air group ,UA)和浓缩PM2.5组(concentrated ambient PM2.5 group, CAP)。每组2个重复,共6个样本。
摘要
环境细颗粒物(PM2.5)对男性生殖健康的影响已引起广泛关注,其损害的具体潜在机制仍不清楚,需要在新的方向进行更多研究。先前研究表明,DNA甲基化在男性生殖发育中起着重要作用,也易受环境影响,然而对于DNA甲基化参与PM2.5诱导的男性生殖毒性作用还没有足够研究。为此,本研究建立一个实时PM2.5暴露模型,揭示了PM2.5暴露可能导致睾丸功能障碍,包括精子发生障碍和类固醇激素功能障碍。睾丸5-甲基胞嘧啶(5mC)全基因组水平降低表明DNA甲基化可能与PM2.5暴露诱导的睾丸损伤有关。进一步的全基因组甲基化分析揭示了睾丸DNA的基因组低甲基化,并在CAP组与UA组和FA组中发现1000多个差异甲基化区域(DMR),表明PM2.5暴露(即使低剂量)可以调控睾丸甲基化。此外,甲基化组和转录组的联合分析鉴定出一些关键甲基化基因和网络,这些基因和网络可能参与精子发生和类固醇激素的合成。关键基因尤其是Cyp11a1和Pax8的睾丸甲基化水平升高,随后表达水平降低可能会损害睾酮和精子生成过程。本研究为DNA甲基化可能参与PM2.5诱导的男性生殖损伤提供了基础知识和新见解。
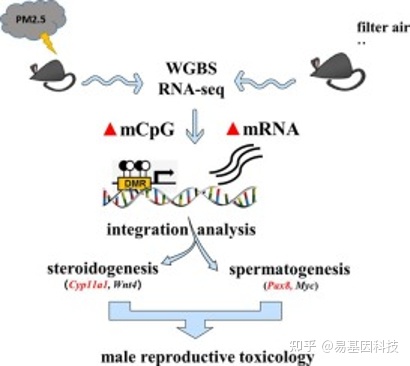
图形摘要
睾丸全基因组DNA甲基化组和转录组的联合分析鉴定了与类固醇生成和精子发生过程相关的关键基因和通路,这可能是PM2.5引起的男性生殖障碍中DNA甲基化调控机制。
研究思路
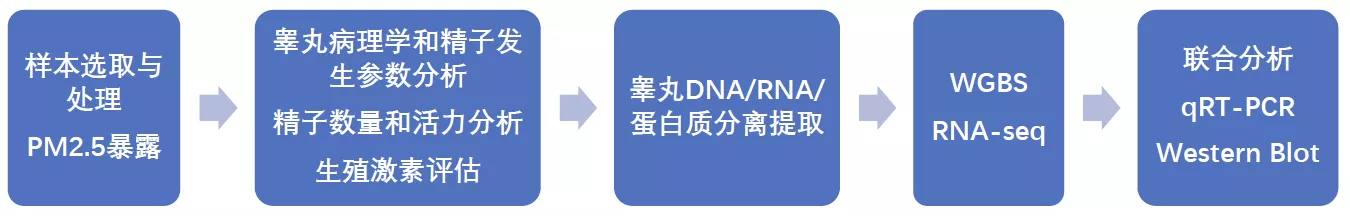
材料方法
8周龄C57BL/6雄性小鼠,过滤空气组(FA),未过滤空气组(UA)和浓缩PM2.5 组(CAP)。FA组为高效颗粒空气过滤器过滤的环境空气,UA组为未经过滤的环境空气,CAP组为细颗粒物(PM2.5)暴露环境空气。
从小鼠眼眶丛中获得血液,称重睾丸和附睾,分别固定在Bouin溶液中进行形态学分析和在液氮中速冻-80°C储存。
从睾丸组织中分离提取RNA、DNA和蛋白质进行全基因组重亚硫酸盐测序 (WGBS)分析、转录组测序(RNA-seq)和Western Blot实验,WGBS为每组构建两个库,每个库包含来自四只小鼠的合并睾丸DNA。
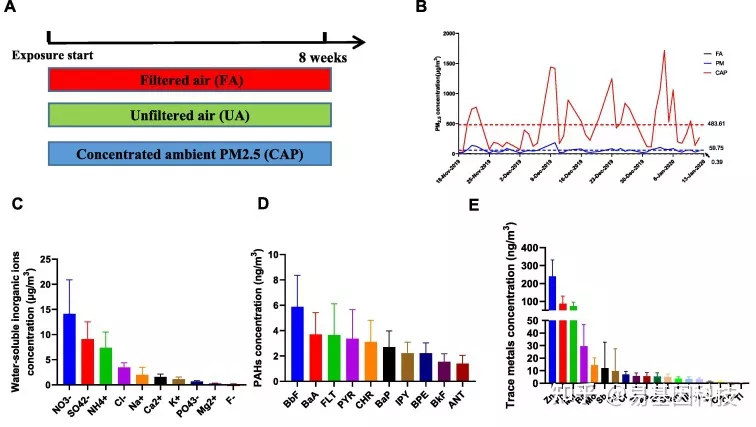
图1:PM2.5暴露表征
(A)PM2.5暴露程序示意图。(B)实验期间各组PM2.5浓度。(C) PM2.5中无机离子水平。(D)PM2.5中多环芳烃浓度。(E)PM2.5中痕量金属(trace metal)浓度。
研究结果
(1)PM2.5暴露导致精子质量下降和精子发生损伤
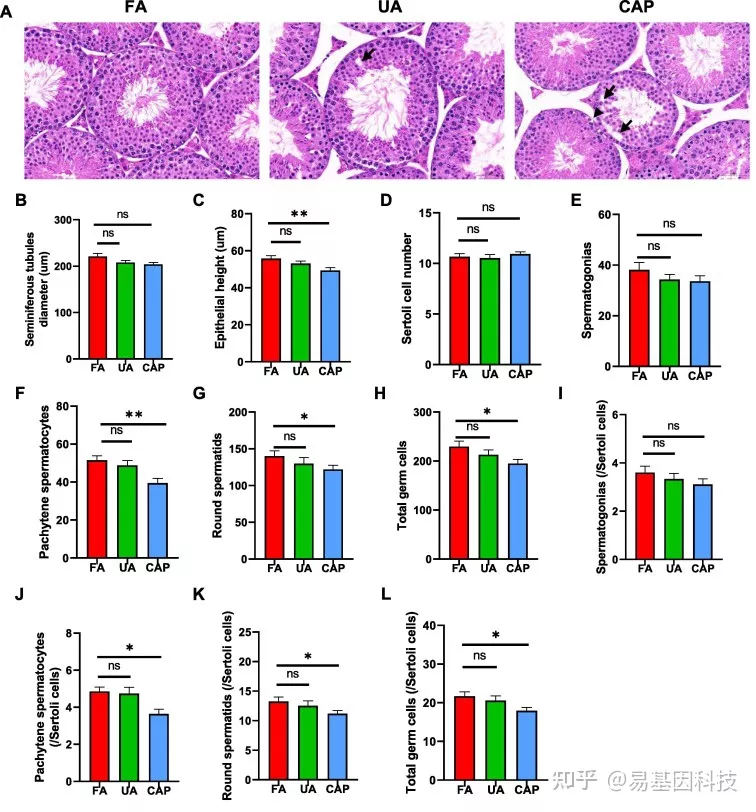
图2:PM2.5暴露导致精子发生障碍
(A)睾丸的典型病理图像。三角形(基底膜中断),黑色箭头(空泡化)。
(B-C)在VII期曲细精管(n=15)中检测曲细精管直径(B)和上皮高度(C)的精子发生参数。
(D-H)每个VII期曲细精管的Sertoli细胞(D)、精原细胞(E)、粗线期精母细胞(F)、圆形精子细胞(G)和总生殖细胞(H)的计数(n=15)。
(I-L)精原细胞/Sertoli细胞比率(I)、粗线期精母细胞/Sertoli细胞比率(J)、圆形精母细胞/Sertoli细胞比率(K)、总生殖细胞/Sertali细胞比率(L)(n=15)。
单向方差分析,然后Tukey多重比较。与FA处理组相比,P<0.05,P<0.01,P<0.001。
(2)PM2.5暴露诱发睾丸类固醇激素功能障碍
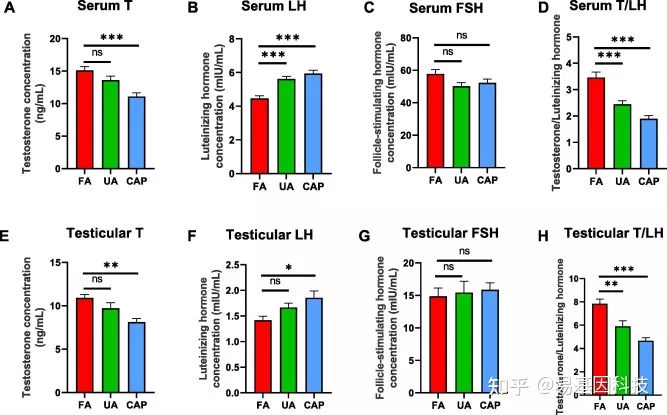
图3:PM2.5暴露诱发睾丸内分泌功能障碍
(A-C)血清中的激素。睾酮(T)(A)、促黄体生成素(LH)(B)、卵泡刺激素(FSH)(C)的水平
(D)血清T/LH在血清中的比值(D)(n=15)。
(E-G)睾丸水平,睾酮(T)(E)、LH(F)、FSH(G)(n=7)。
(H) 睾丸T/LH比值(n=7)。
(3)PM2.5暴露诱导睾丸全基因组低甲基化
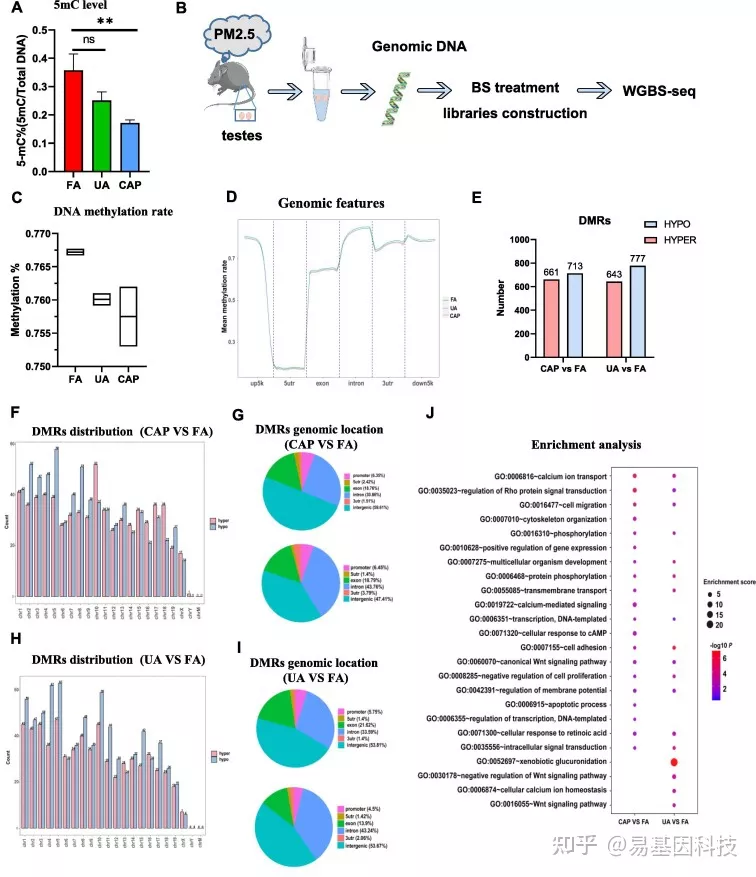
图4:PM2.5暴露改变了睾丸中的甲基化模式
(A) ELISA检测到的睾丸DNA的全基因组5mC甲基化水平。
(B) 基因组甲基化测序示意图。
(C) 通过WGBS检测睾丸基因组中所有CpG位点的全基因组DNA甲基化水平。
(D) 功能基因组区域mm10内5mC相对密度的基因组特征。
(E) PM2.5暴露组与FA组DMR鉴定。
(F和H) CAP组(F)和UA组(H)染色体上的显著DMR。
(G和I) CAP组(G)和UA组(I)中鉴定的DMR基因组位置(启动子、5’UTR、外显子、内含子、3’UTR、基因间)(上:高DMR;下:低DMR)。
(J) DMR相关基因(DMG)生物过程的富集分析。点的颜色表示P值,点的大小是给定生物过程中的富集分数。
(4)基于DMR的功能相关基因
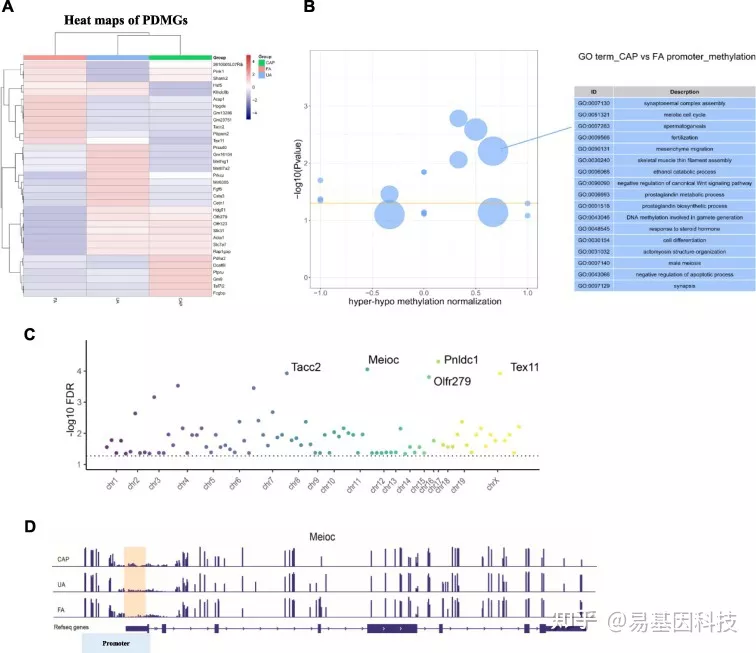
图5:PM2.5诱导的启动子相关DMR变化表征
(A) FA组、UA组和CAP组中启动子连接的DMR相关基因(PDMG)热图。块的颜色表示相对甲基化水平。红色表示高甲基化,蓝色表示低甲基化。
(B) 与FA组相比,CAP组中PDMGs的最高生物过程(top biological process)气泡图。每个圆圈表示不同的生物过程,大小表示富集-sore。X轴表示总差异基因中高甲基化和低甲基化基因数量差异比例。气泡越向右,表示通路中高甲基化基因比例就越高。
(C) 启动子区域的DMG以及染色体位置的曼哈顿图。X轴代表染色体。Y轴是-log10 FDR。
(D) DNA甲基化peaks可视化Meioc基因座。橙色表示DMR区域。
(5)PM2.5暴露破坏了睾丸的转录组特征
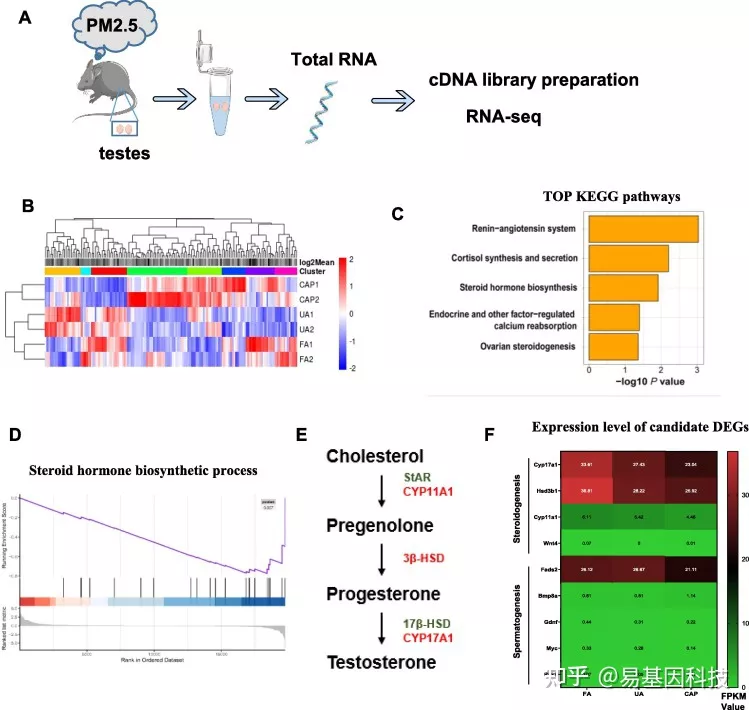
图6:PM2.5暴露破坏睾丸的转录组学特征
(A) 实验设计图。
(B) FA组、UA组和CAP组中DEG热图。
(C) DEG的KEGG分析。
(D) FA组和CAP组中类固醇激素生物合成过程基因组的GSEA分析。
(E) 睾酮生物合成通路分子图。
(F) 与类固醇生成和精子发生相关的DEGs热图。块的颜色表示每组的基因表达水平。
(6)DNA甲基化组和转录组联合分析,鉴定与PM2.5暴露相关的关键甲基化基因
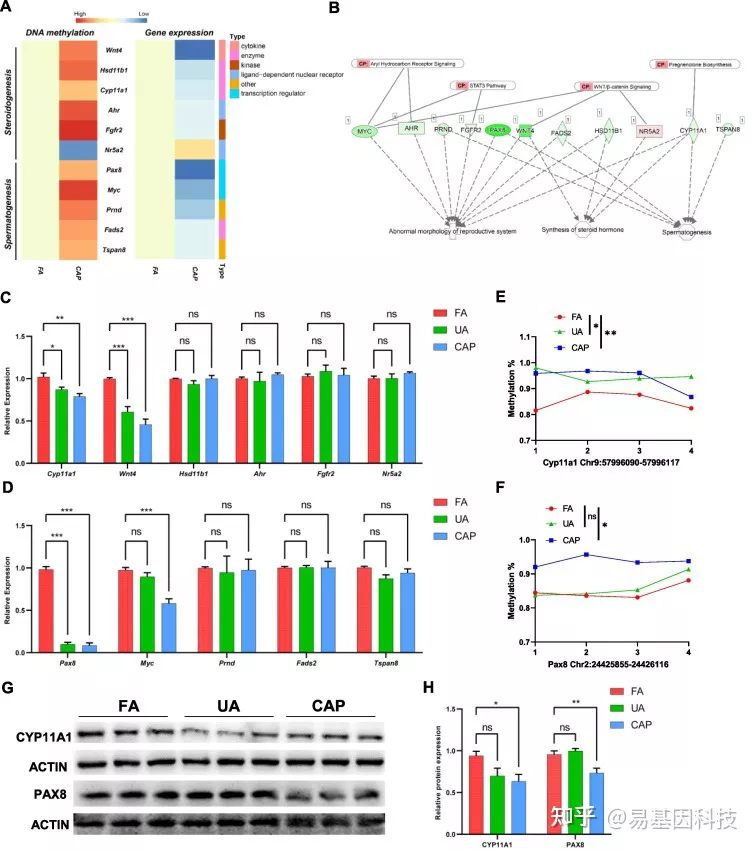
图7:WGBS和RNA-seq图谱联合分析揭示了PM2.5诱导的睾丸损伤中候选表观遗传基因和网络失调
(A)与类固醇生物合成和精子发生相关的候选功能基因的启动子甲基化和表达水平热图。
(B) 参与生殖功能的候选基因网络。
(C) RT-PCR验证睾丸中候选类固醇生成相关基因的mRNA表达水平(n=6)。
(D) RT-PCR验证睾丸中候选精子发生相关基因的mRNA表达水平(n=6)。
(E) Cyp11a1基因DMR内代表性CpG甲基化谱。
(F) Pax8基因DMR内代表性CpG甲基化谱。
(G) western blot分析睾丸候选基因的蛋白表达水平。
(H) CYP11A1和PAX8的蛋白质定量分析(n=6)。
结论:
本研究表明PM2.5暴露可导致睾丸功能障碍,包括精子发生和类固醇激素功能障碍。作者揭示了PM2.5可以改变睾丸的甲基化水平,表明DNA甲基化可能参与PM2.5诱导的男性生殖损伤过程。睾丸全基因组DNA甲基化组和转录组的联合分析鉴定了与类固醇生成和精子发生过程相关的关键基因和通路,这可能是PM2.5导致男性生殖障碍的DNA甲基化调控机制。本研究可能为PM2.5诱导男性生殖毒性的潜在表观遗传机制提供重要信息和新视角。
关于全基因组重亚硫酸盐测序(WGBS)
全基因组重亚硫酸盐甲基化测序(WGBS)可以在全基因组范围内精确的检测所有单个胞嘧啶碱基(C碱基)的甲基化水平,是DNA甲基化研究的金标准。WGBS能为基因组DNA甲基化时空特异性修饰的研究提供重要技术支持,能广泛应用在个体发育、衰老和疾病等生命过程的机制研究中,也是各物种甲基化图谱研究的首选方法。
易基因全基因组甲基化测序技术通过T4-DNA连接酶,在超声波打断基因组DNA片段的两端连接接头序列,连接产物通过重亚硫酸盐处理将未甲基化修饰的胞嘧啶C转变为尿嘧啶U,进而通过接头序列介导的 PCR 技术将尿嘧啶U转变为胸腺嘧啶T。
应用方向:
WGBS广泛用于各种物种,要求全基因组扫描(不错过关键位点)
- 全基因组甲基化图谱课题
- 标志物筛选课题
- 小规模研究课题
技术优势:
- 应用范围广:适用于所有参考基因组已知物种的甲基化研究;
- 全基因组覆盖:最大限度地获取完整的全基因组甲基化信息,精确绘制甲基化图谱;
- 单碱基分辨率:可精确分析每一个C碱基的甲基化状态。
易基因科技提供全面的DNA甲基化研究整体解决方案,技术详情了解请致电易基因0755-28317900。
参考文献:Zhang Z, Wang J, Shi F, Li Y, Zou P, Tang Y, Liu C, Wang Y, Ling X, Sun L, Liu C, Zhang Y, Gao F, Chen Q, Ao L, Han F, Liu J, Cao J. Genome-wide alternation and effect of DNA methylation in the impairments of steroidogenesis and spermatogenesis after PM2.5 exposure. Environ Int. 2022 Sep 24;169:107544.
往期回顾:
WGBS跟RRBS该如何选?表观DNA甲基化和转录调控结果关联思路
项目文章 | 90天见刊,易基因m6A RNA甲基化(MeRIP)+转录组组学研究