今天我们要给大家分享的是一个实用的差异分析工具——GEO2R。不需要语言基础,直接点点点就可以得到自己想要的数据分析结果。
GEO2R是GEO数据库中的一个在线分析工具,那么在介绍GEO2R之前,我们先来给大家简单介绍一下GEO数据库。
GEO数据库全称GENE EXPRESSION OMNIBUS,是由美国国立生物技术信息中心NCBI创建并维护的基因表达数据库。它创建于2000年,收录了世界各国研究机构提交的高通量基因表达数据,主要储存芯片、二代测序以及其他高通量测序数据的一个数据库。目前大约百分之九十已经发表的论文,论文中涉及到的基因表达检测的数据都可以通过这个数据库中找到。
1、点击 https://www.ncbi.nlm.nih.gov/geo/ 进入网站。
2、点击上图红色圈处“series”,得到下图。
(Series代表数据集,Samples代表样本,Platforms 代表平台)
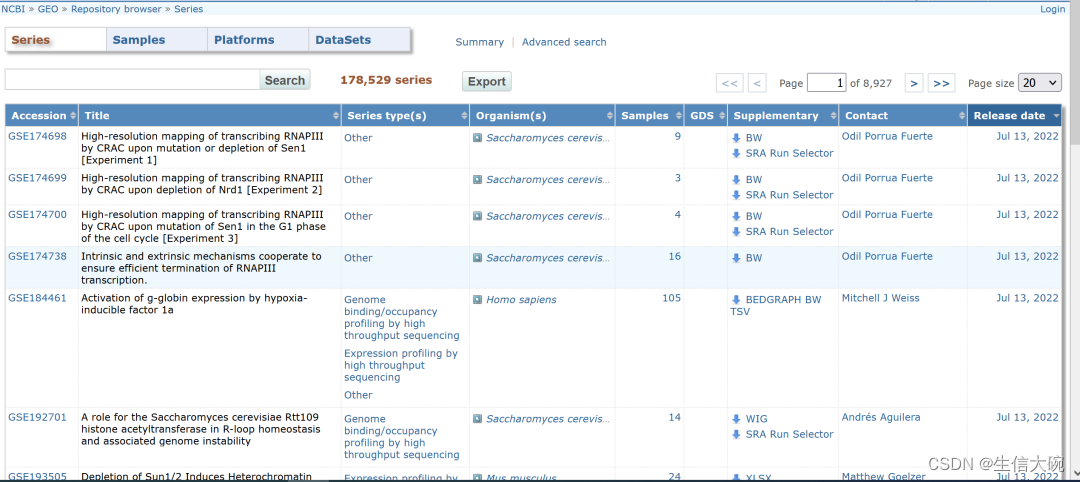
3、下面我们以肝癌的表达数据为例,在Search方框内输入 HCC,点击搜索后,我们选择数据集GSE166163,点击下图画圈处。
4、点击下图Analyze with GEO2R。
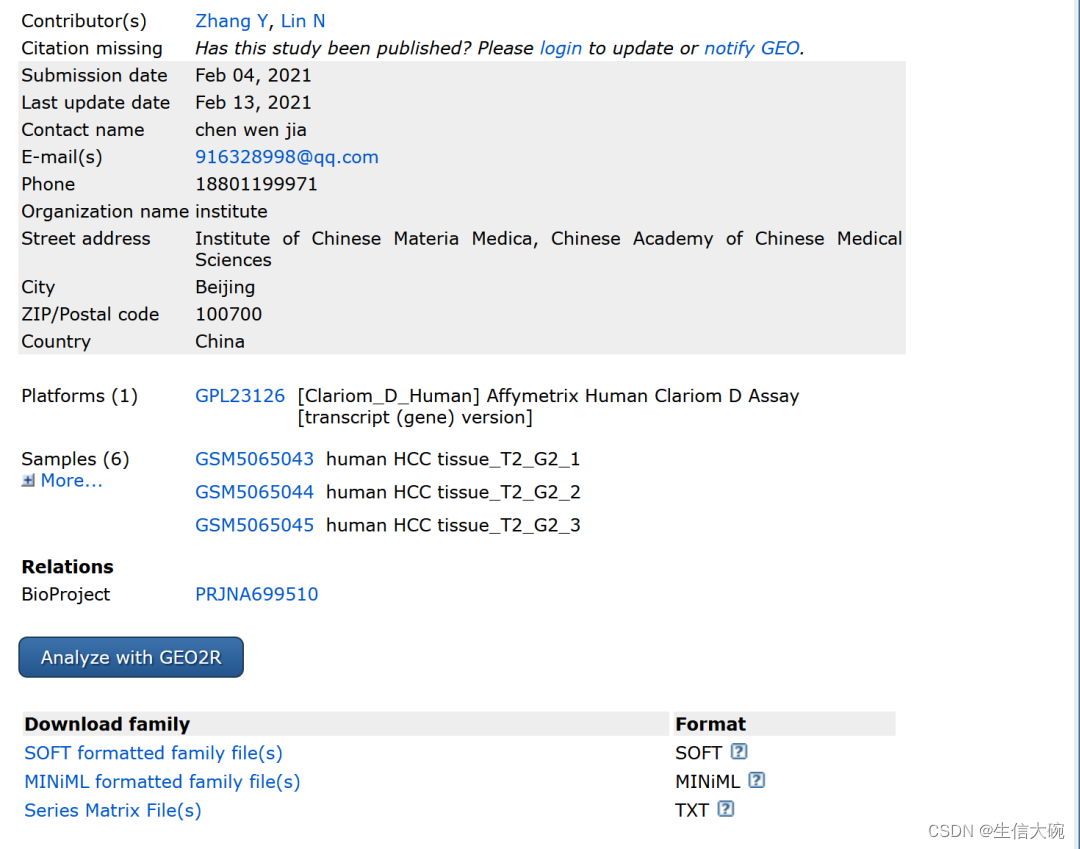
5、得到如下图所示界面,我们需要点击定义分组。
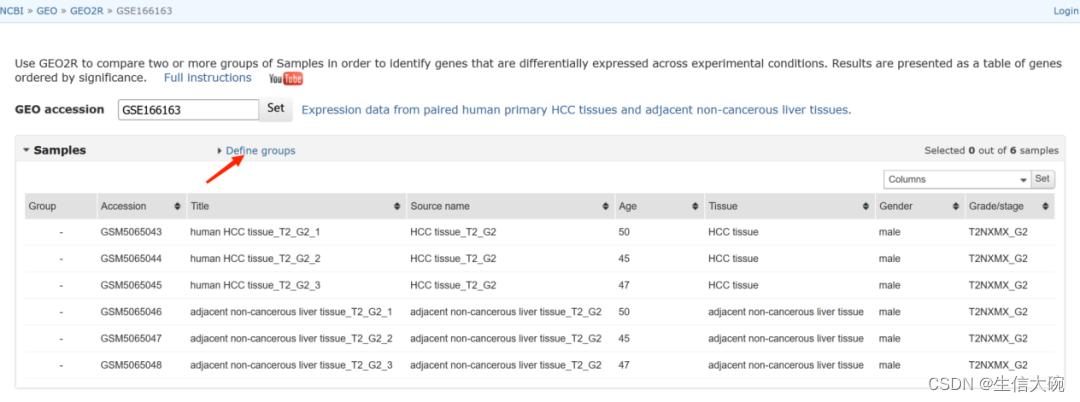
6、在定义分组方框内输入分组的名称,然后直接点击Enter即可。
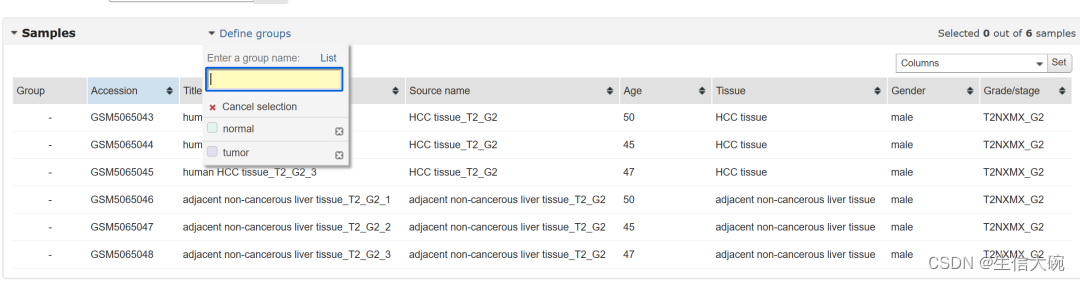
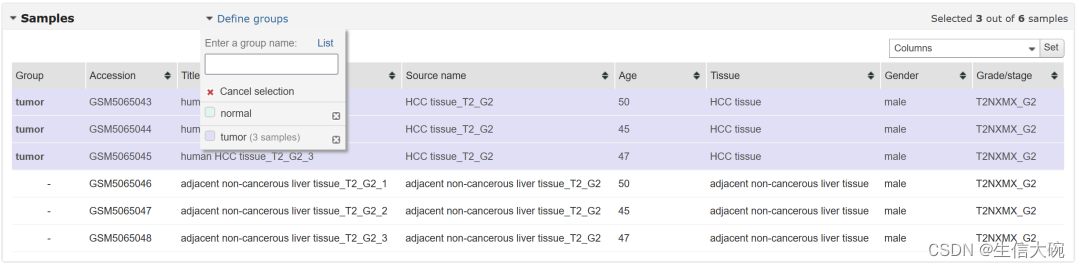
7、我们先勾选下方肿瘤组三个样本,鼠标长按滑动即可,当三个样本出现一致颜色后,点击上方肿瘤组即可如下图所示,tumor组则出现了三个样本。
(正常组也一样)
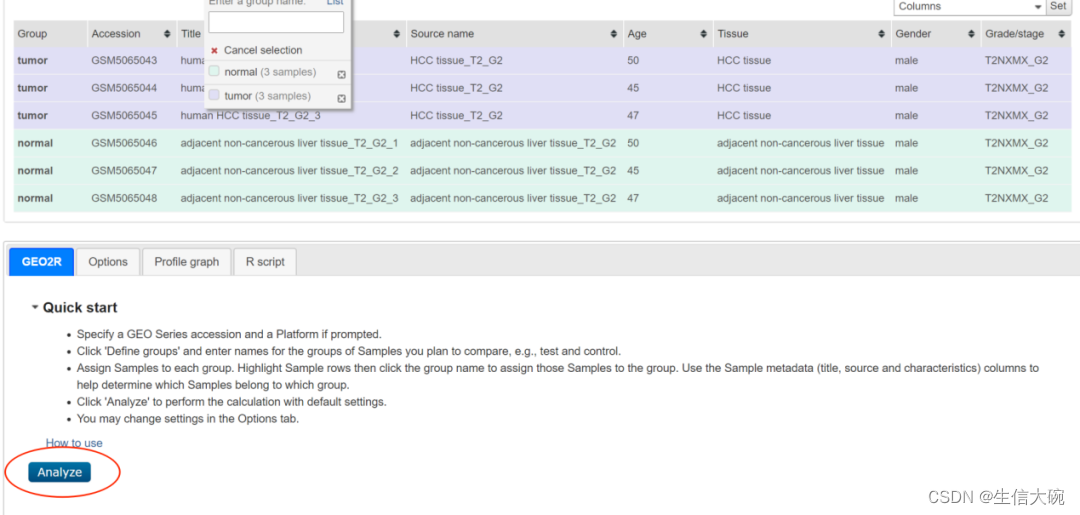
8、分组信息定义好后,直接点击下方Analyze,即可得到差异分析的结果和图。
(这一步可能会因为网络原因或者分析的数据较大会花费一些时间)
9、下图是我们得到的癌症与正常组差异分析的结果。图上半部分是我们得到的差异分析的火山图,下半部分是差异分析结果的表格。
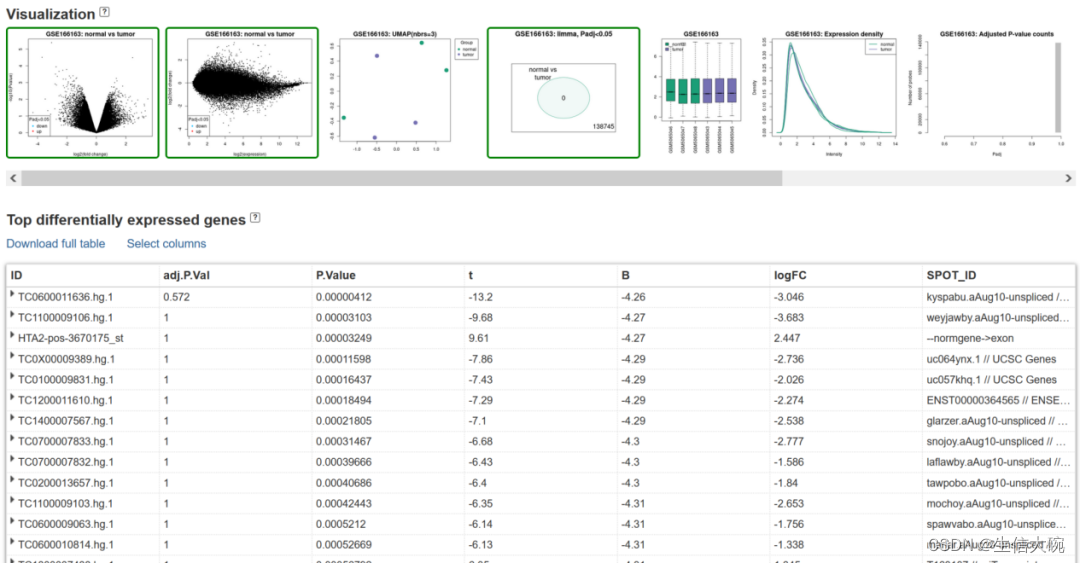
小结
为什么有些数据集没有GEO2R的这个工具显示呢?
为什么这个火山图的可视化界面没有颜色差异呢?
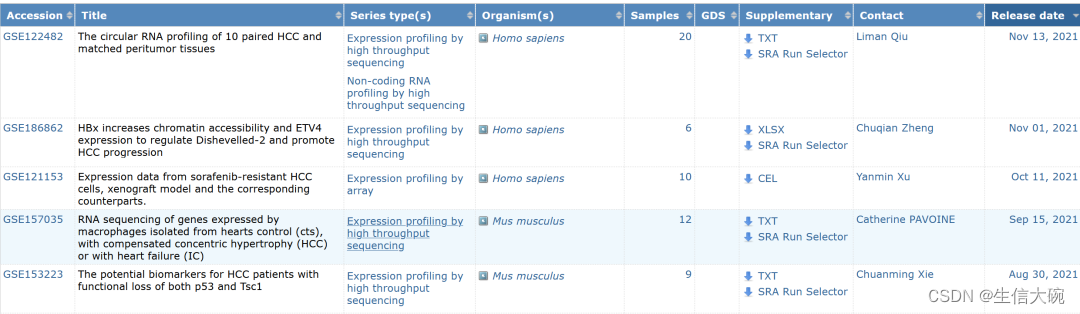
1、GEO2R分析工具是基于R语言写的分析工具,R语言处理不同数据时,所需要的脚本不同。GEO收录了很多不同的数据类型,如芯片数据和二代测序数据,而GEO2R只能分析芯片数据,也就是Series type列以array结尾的数据类型。而一般以high throughput sequencing结尾的数据类型二代测序。
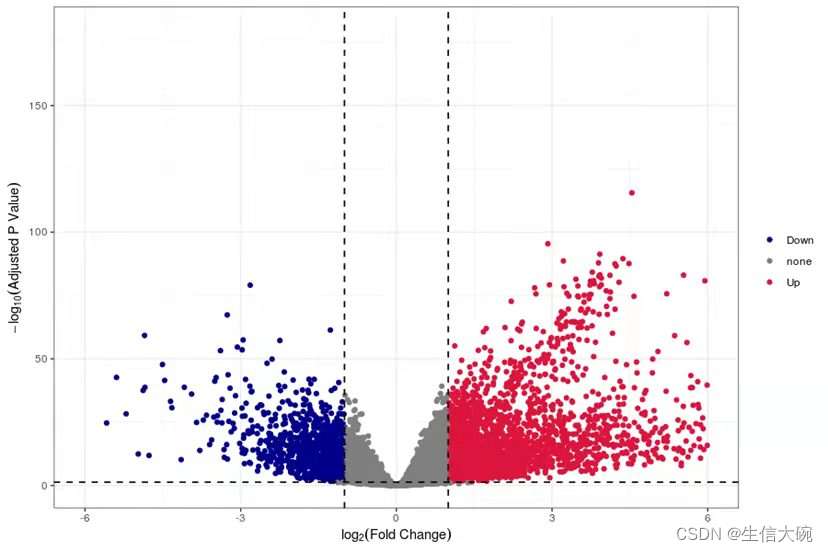
2、关于火山图的颜色,我们一般得到的火山图如下图,每个点代表一个基因,红色代表高表达基因,蓝色代表低表达基因,中间灰色代表没有统计学意义的基因。
3、那这么说来,我们分析的这个数据集GSE166163没有意义吗?
其实不是的。GEO2R分析是是以调整后的P值计算统计学意义的,而当我们用到这样的数据的时候可以用P值来筛选,这样也可以得到一些有意义的基因。
好了,今天的分享就到这里结束啦,如果你想学习更多的生物信息学知识,欢迎关注微信公众号——生信大碗。
