目录
NIFTI文件结构
NIFTI 格式,是一种用于存储和交换医学成像数据的文件格式,特别适用于神经影像学领域。NIFTI文件通常有两个扩展名:.nii(用于图像数据)和 .hdr(用于头文件,包含有关图像的元数据)。这两个文件是成对出现的,.hdr 文件包含有关图像数据的详细信息,如尺寸、方向、数据类型等,而 .nii 文件则包含实际的图像数据。
读取NII文件
在医学图像上,nii格式通常用于存储3D的图像,其中包含了多个2D切片的集合。这些切片按顺序排列,共同构成了3D图像数据。如果想要可视化NII文件,一般会将其切片转换为2D图像或直接使用ITK-SNAP软件查看。由于目前我所训练的图像数据均为2D,因此我采用了纵向切片的方法处理,但在此之前需要查看图像数据是否有损坏。
ITK-SNAP安装
官网链接:http://www.itksnap.org/pmwiki/pmwiki.php?n=Downloads.SNAP4
最新版本:https://sourceforge.net/projects/itk-snap/
这里根据自己的电脑配置下载,后面正常安装即可
使用方法
打开ITK-SNAP软件 - > file - > Open Main Image
下面显示的就是一个数据信息保存完整的nii文件
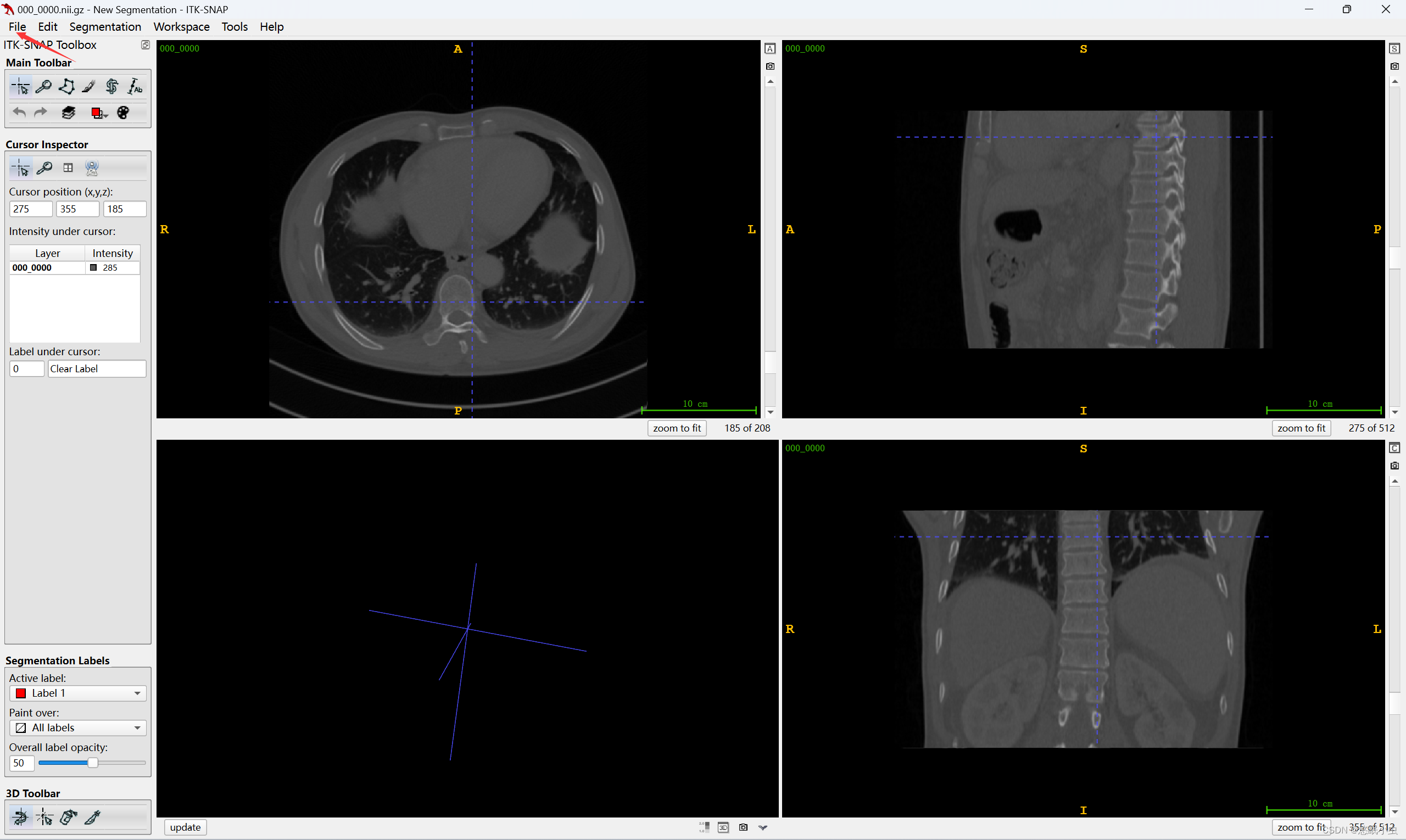
这个只是很简单的查看一个 .nii 文件是否能够正常显示,ITK-SNAP软件对于3D图像的处理很有帮助,但我对3D图像数据的处理方式并不熟悉,因此并未深入探讨,如果有想要深入了解该软件的使用方法,请移步ITK-Snap 处理医疗影像。
NII转PNG
由于目前我所接触训练数据都是2D图像,而生物医学工程竞赛中提供的数据集大部分都是这种 nii 格式的文件,因此在遇到这种 nii 文件时我都会先将其转换为PNG格式的图像在进一步处理。这里给大家提供一个第9届生物医学工程竞赛的题目:基于CT图像的肝细胞癌肿瘤区域分割
百度网盘链接:https://pan.baidu.com/s/1KWSAJyP0MdwblihkbrFHOg
提取码:hwj3
网盘里面包含官方提供的数据集以及题目文档,感兴趣的可以跟着一起尝试一下

一个数据集通常包含图像和标签,二者一一对应,这里两个都需要转换
import os
import nibabel as nib
import imageio
import numpy as np
from tqdm import tqdm
def nii_to_image(filepath, imgfile):
filenames = os.listdir(filepath) # 读取nii文件夹
with tqdm(total = len(filenames)) as pbar:
for f in filenames:
if f[-7:] != ".nii.gz":
continue
img_path = os.path.join(filepath, f)
img = nib.load(img_path) # 读取nii
img_fdata = img.get_fdata(dtype=np.float32) # 读取为float32
fname = f.replace('.nii.gz', '') # 去掉nii的后缀名
img_f_path = os.path.join(imgfile, fname)
# 创建nii对应的图像的文件夹
if not os.path.exists(img_f_path):
os.mkdir(img_f_path) # 新建文件夹
# 将归一化的浮点数数据转换为8位整数数据
img_fdata = (img_fdata * 255).astype(np.uint8)
# 开始转换为图像
(x, y, z) = img.shape
for i in range(z): # z是图像的序列
slice = img_fdata[:, :, i] # 选择哪个方向的切片都可以
# 保存图像,使用PNG格式
imageio.imwrite(os.path.join(img_f_path, '{}.png'.format(i)), slice)
pbar.update(1)
file_name = r"E:\Desktop\生医竞赛:CT肝细胞\liver_tumor_segmentation\imagesTr"
img_path = r"E:\Desktop\0"
nii_to_image(file_name, img_path)此处使用纵向切片的方法(可以按需要修改为其它方向),对每一个3D图像切片为2D图象并保存为PNG格式(保存的图像格式也可以改为JPG或其它格式)
这里 file_name 是包含NII文件的文件夹路径,img_path 是输出图像文件夹的路径,自己修改即可
这相当于对一个具有体积的物体进行切片,因此每一个3D图像都会得到数量不一的2D图像
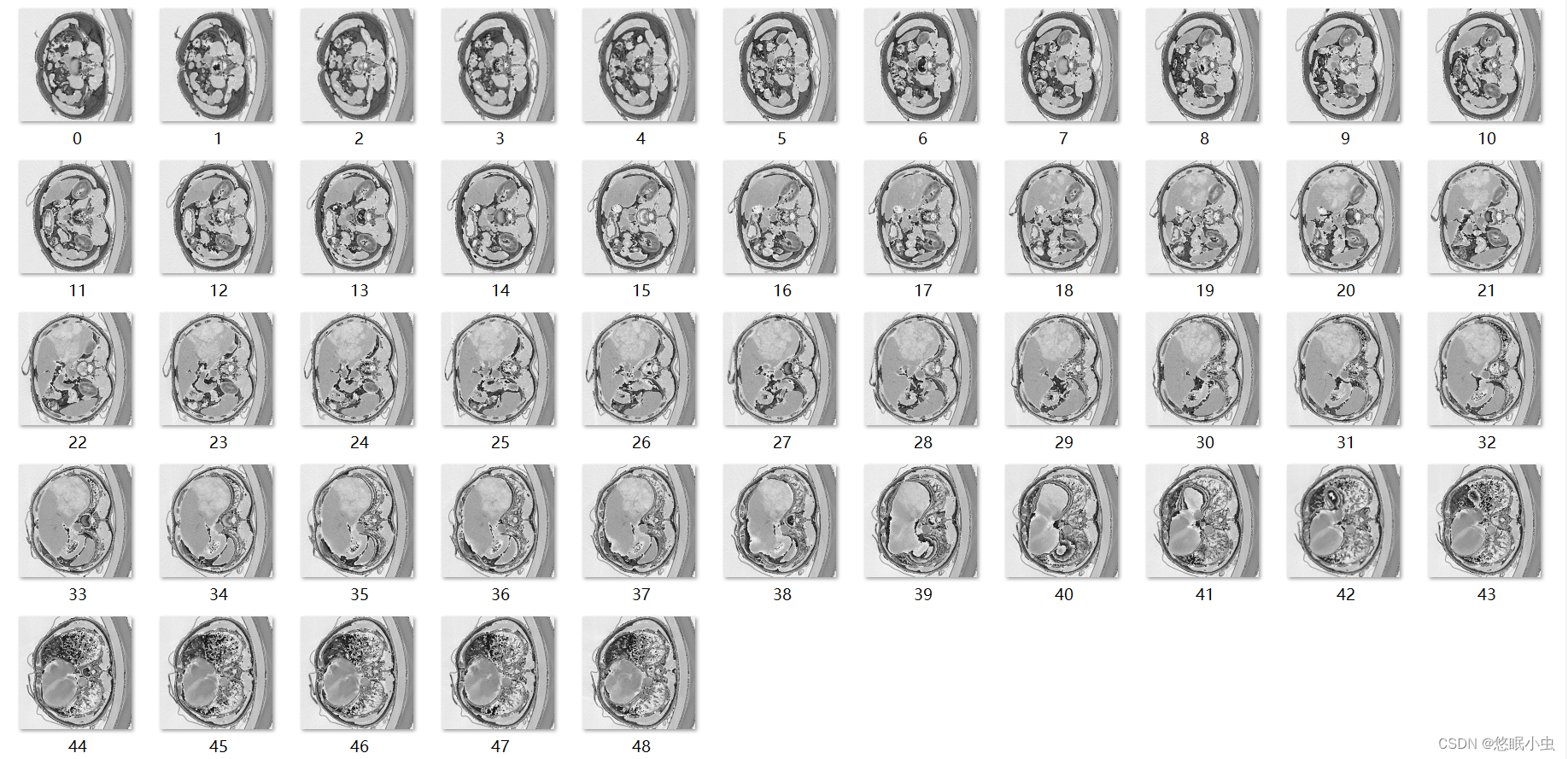
生成的image图像
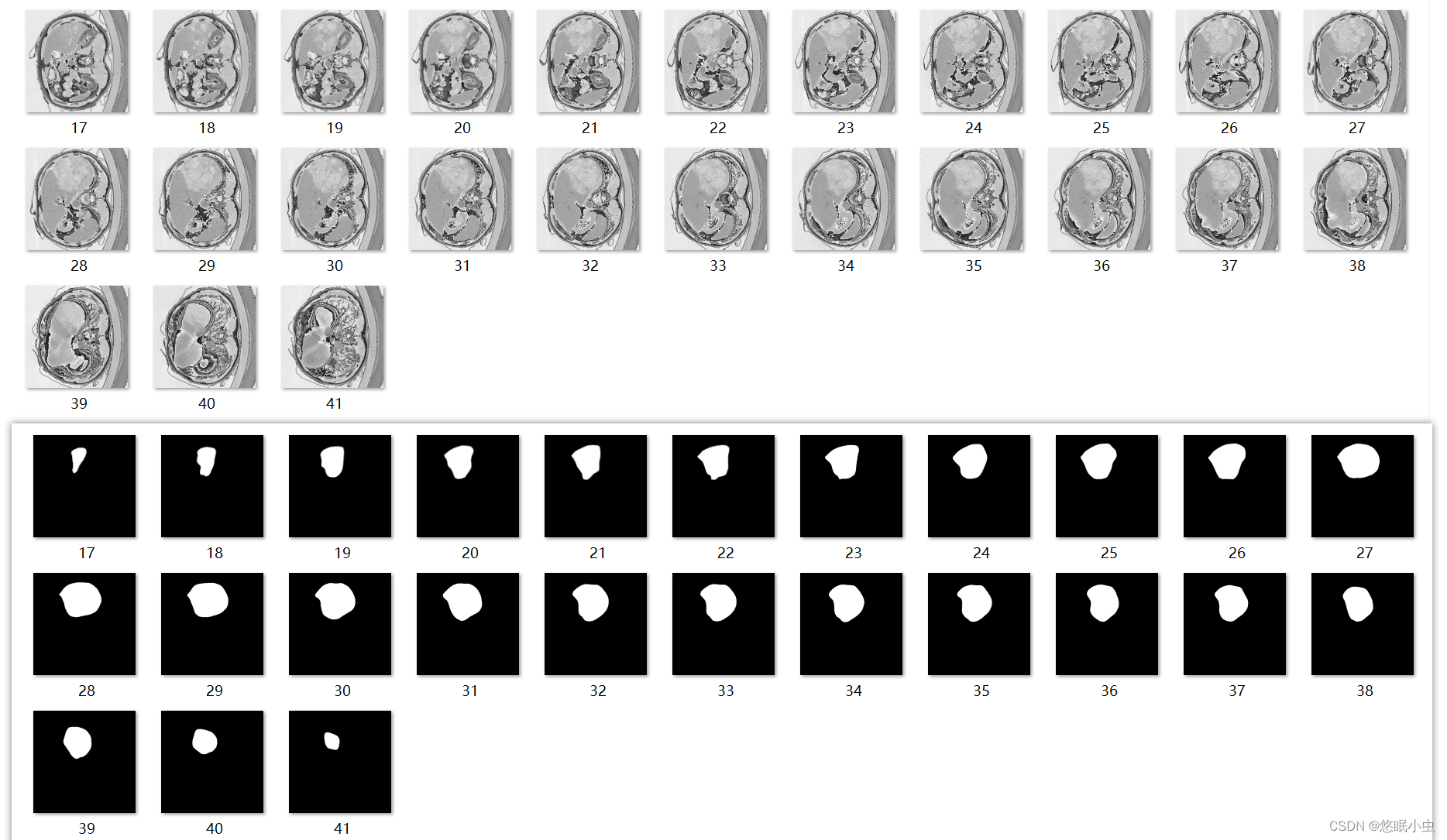
生成的label图像

从label中可以看到有些图像是全黑的,说明这些label对应的image图像中并没有病变的区域,而这些图像是我们训练所不需要的,因此需要去除这些无效的image图像以及对应的label,并且由于切片操作使得图像数量庞大,手动去除的话肯定是非常耗时的,因此这里也提供去除无效image图像方法,但这个有很大的弊端:需要手动去除label里面全黑的图像。
import os
import shutil
def remove_unmatched_image_subfolders(image_dir, label_dir):
label_subfolders = {os.path.basename(f) for f in os.listdir(label_dir) if os.path.isdir(os.path.join(label_dir, f))}
for image_subfolder in os.listdir(image_dir):
image_subfolder_path = os.path.join(image_dir, image_subfolder)
# 检查image中的子文件夹是否以_0000结尾,如果是,去除_0000后再进行匹配
if image_subfolder.endswith("_0000"):
image_subfolder_base = image_subfolder[:-5] # 去除_0000
# 如果image中的子文件夹的基准名称(去除_0000后)不在label的子文件夹列表中,则删除
if image_subfolder_base not in label_subfolders:
print(f"Deleting unmatched subfolder in image: {image_subfolder}")
shutil.rmtree(image_subfolder_path)
# 设置test文件夹中image和label的路径
test_folder_path = r'E:\Desktop\example'
image_folder_path = os.path.join(test_folder_path, 'image')
label_folder_path = os.path.join(test_folder_path, 'label')
# 删除image中与label不匹配的子文件夹
remove_unmatched_image_subfolders(image_folder_path, label_folder_path)
print("Cleanup completed.")去除后的效果
这样上面得到的数据集就可以用于常规的神经网络训练咯。其实3D的图像数据也可以直接用于网络训练,但是目前博主并未进行深入的学习,因此处理这种数据时都会先将其转换为2D在进行模型训练。